Ein Beispiel
Betrachten wir die dreidimensionale Raumstruktur (Tertiärstruktur) eines kleinen Proteins; in der Wikipedia fand sich vor Jahren ein schönes Beispiel, das als public-domain-Bild veröffentlicht wurde:
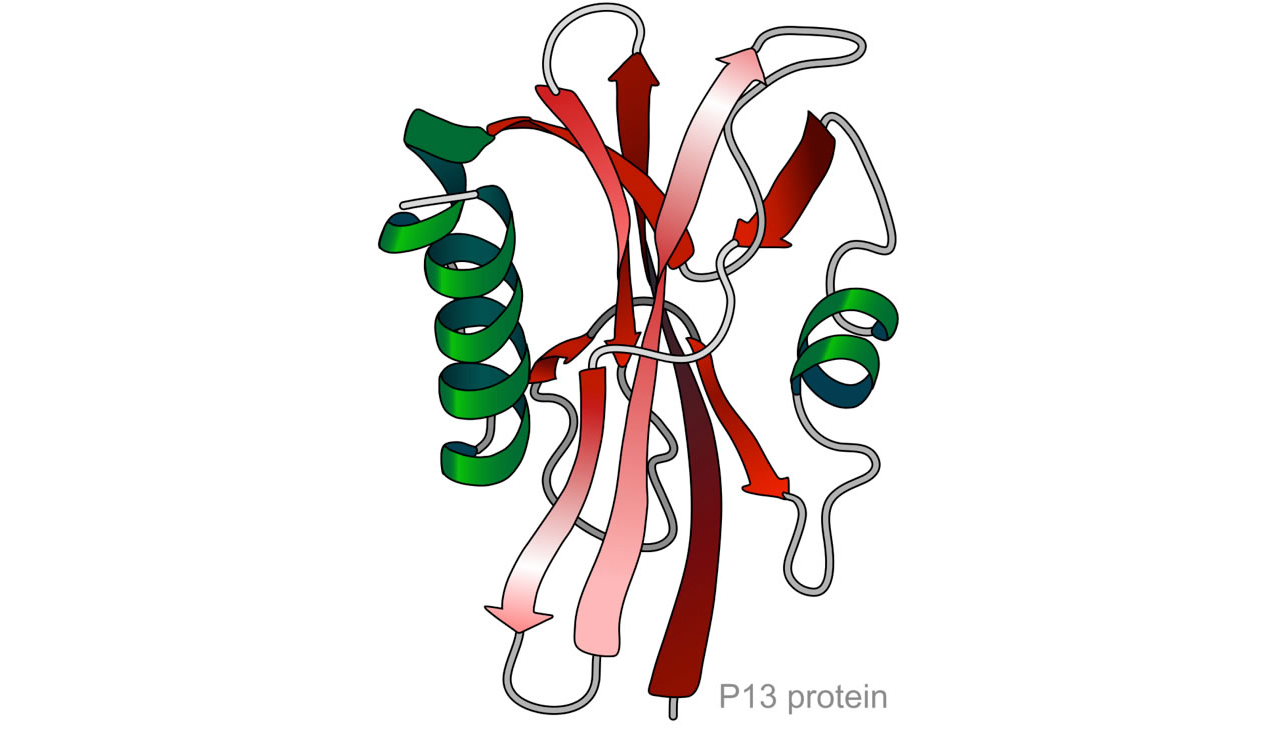
Die Tertiärstruktur eines kleinen Proteins
Quelle: Wikipedia, Artikel "Protein structure", abgerufen im Mai 2013, Autor: nicht mehr bekannt, Lizenz: public domain.
In dieser Tertiärstruktur kann man verschiedene Bereiche erkennen, zwei spezielle Faltungsmuster und einen unstrukturierten Bereich:
- Alpha-Helices (die grün gezeichneten Spiralen)
- Beta-Faltblätter (die rot gezeichneten Pfeile)
- ungeordnete Bereiche (die grau gezeichneten Schlingen)
Die α-Helix- und β-Faltblatt-Bereiche bezeichnet man auch als Sekundärstrukturen des Proteins.
Sekundärstrukturen
Geordnete Faltungsmuster innerhalb der Raumstruktur eines Proteins. Man unterscheidet α-Helix- und β-Faltblatt-Bereiche.
Die α-Helix wurde zuerst entdeckt
Beide Sekundärstrukturen wurden in den 60er Jahren des letzten Jahrhunderts entdeckt, und zwar bei der Untersuchung von Haaren und Seide [2]. Aber schon vorher, nämlich 1951, postulierten Linus Pauling und Robert Corey die Existenz von Helices und Faltblättern in Polypeptiden [3] als energetisch günstigere Strukturen.
Beide Strukturen bilden sich quasi "von alleine" aus, teils schon während der Translation des Proteins. Ca. ein Jahr nach der Entdeckung der Helix-Struktur wurde dann auch die Faltblatt-Struktur entdeckt.
Eine Peptidkette besteht aus hydrophilen Aminosäuren wie Arginin oder Glutaminsäure und aus hydrophoben Aminosäuren wie Alanin oder Valin. Hydrophobe Aminosäuren "meiden" normalerweise den Kontakt mit Wasser, weil sie durch diesen Kontakt die energetisch sehr günstigen Wasserstoffbrücken-Bindungen zwischen den Wasser-Molekülen zerstören würden (hydrophober Effekt). Für eine lange Kette von Aminosäuren ist es daher energetisch günstiger, sich zusammenzufalten. Und zwar so, dass die hydrophoben Aminosäuren vor dem Kontakt mit dem Wasser geschützt sind.
Wenn sich eine Peptidkette aufgrund dieses hydrophoben Effekts zu einer Helix oder einem Faltblatt zusammenfaltet, so bilden sich Wasserstoffbrücken-Bindungen zwischen nicht-benachbarten Aminosäuren aus. Schauen wir uns noch einmal die Peptidbindungen innerhalb eines Proteins an:
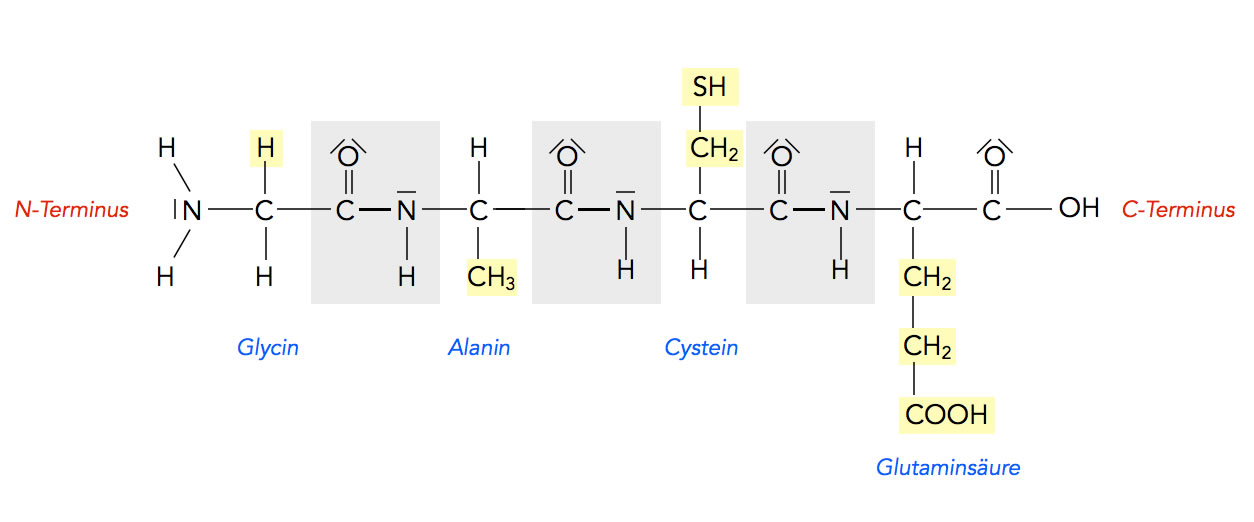
Peptidbindungen innerhalb eines Tetrapeptids
Autor: Ulrich Helmich, Lizenz: Public domain
Das obige Tetrapeptid enthält drei solcher Peptidbindungen. Das Tetrapeptid ist noch zu kurz, um sich "sinnvoll" zu falten.
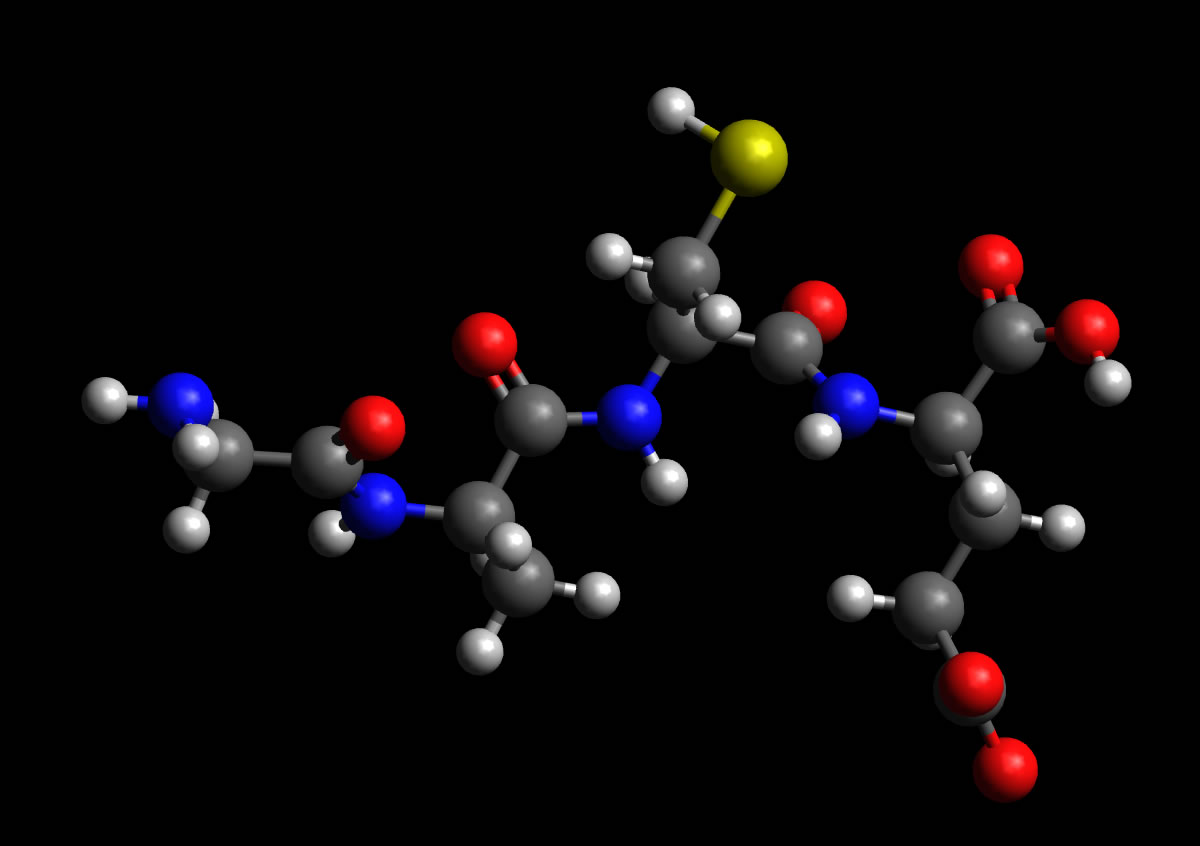
Kugel-Stab-Modell des Tetrapeptids aus Abbildung 2
Autor: Ulrich Helmich, Lizenz: Public domain
Mit der Software "Avogadro" habe ich das Tetrapeptid aus Abbildung 2 einmal nachgebaut, anschließend habe ich die Funktion zur Berechnung der energetisch günstigsten Struktur laufen lassen, und das Ergebnis dieser Berechnung ist in der Abbildung 3 zu sehen. Man kann gut sehen, dass hier keine lineare Kette vorliegt, sondern man kann schon eine Art "Faltung" erkennen - aber natürlich noch keine Helix- oder Faltblatt-Struktur.
Aber kommen wir zurück zu der "einfachen" Zeichnung eines Peptids.
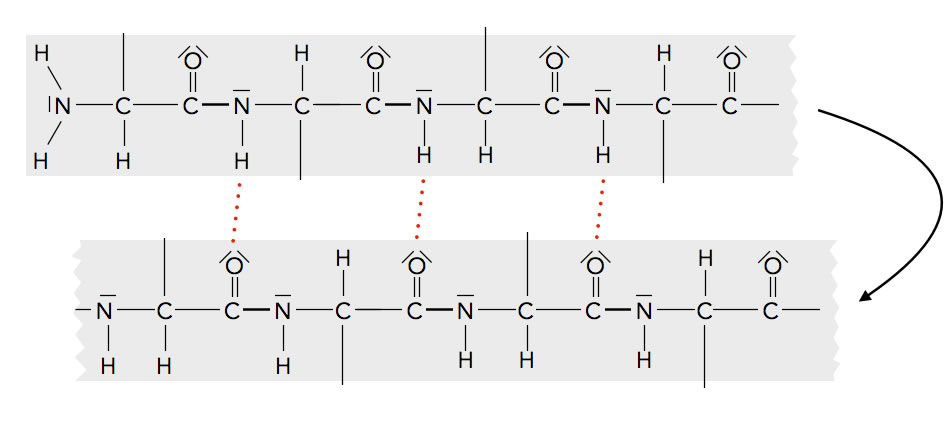
H-Brücken innerhalb eines Peptids
Autor: Ulrich Helmich, Lizenz: Public domain
In dieser stark vereinfachten Zeichnung kann man sehen, wie sich Wasserstoffbrücken-Bindungen zwischen nicht-benachbarten Aminosäuren eines Peptids bilden können. Die Seitenketten wurden der Übersicht wegen einfach mal weggelassen. Wasserstoffbrücken-Bindungen (kurz: H-Brücken) gehören zu den schwachen chemischen Bindungen, sie sind also viel schwächer als kovalente Bindungen. Aber trotzdem, wenn viele solcher H-Brücken gleichzeitig wirken, hat das schon erhebliche Auswirkungen auf die Struktur eines Proteins.
Die Alpha-Helix
Eine α-Helix bildet sich, wenn sich eine Peptidkette in sich selbst verdreht, um einen energetisch günstigeren Zustand einzunehmen. Dabei bilden sich H-Brücken zwischen jeder vierten Peptidbindung.
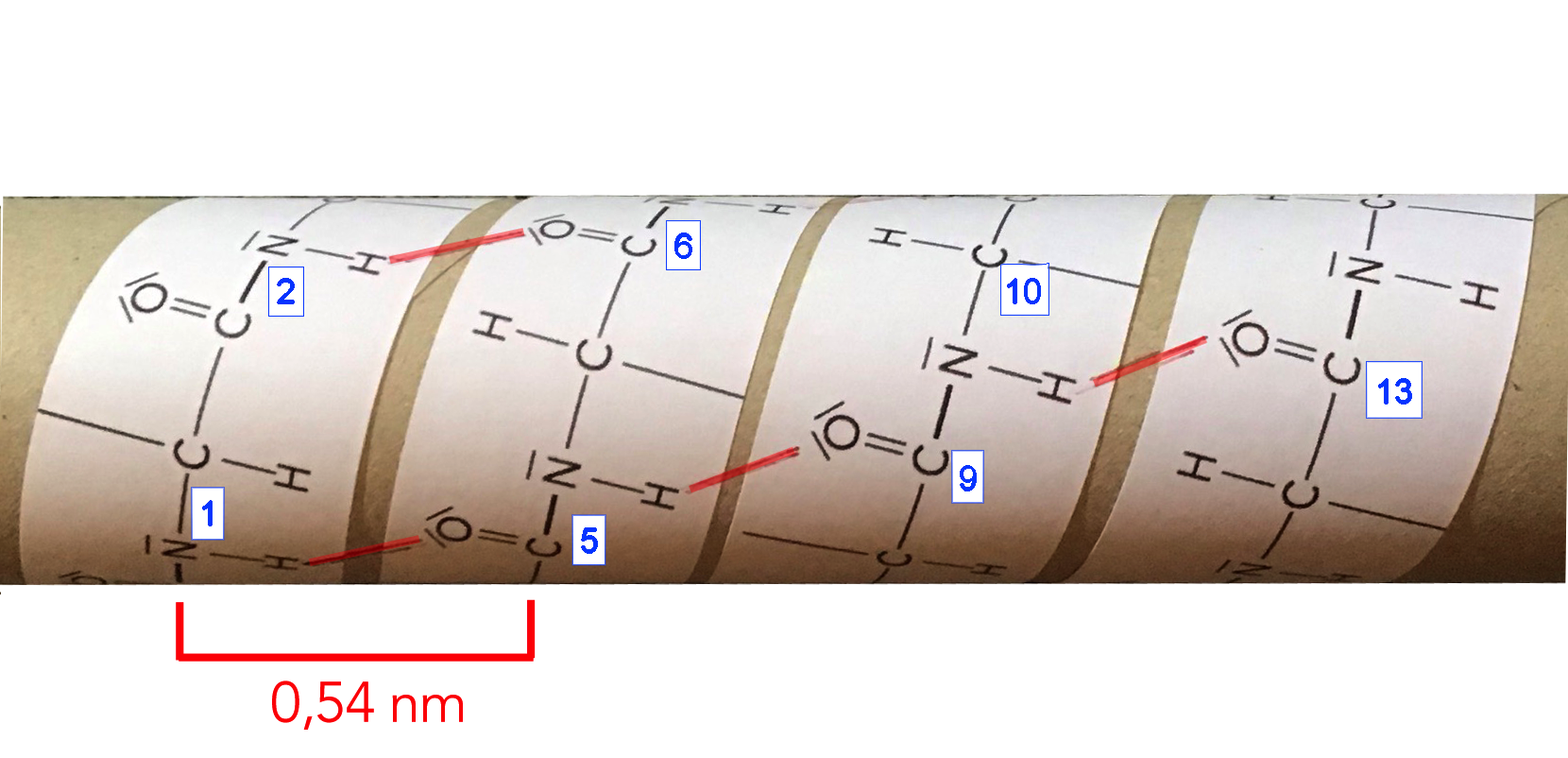
Eine selbst gebastelte alpha-Helix
Autor: Ulrich Helmich, Lizenz: Public domain
Für diese Abbildung habe ich einfach mal ein langes Peptid (ohne Seitenketten) ausgedruckt, ausgeschnitten und auf eine leere Haushaltspapierrolle geklebt. Die H-Brücken habe ich dann in Rot eingezeichnet. Wenn man nun die Aminosäuren abzählt, stellt man fest, dass die 1. Aminosäure mit der 4. verbunden ist, die 2. Aminosäure mit der 5., die 3. Aminosäure mit der 6. und so weiter.
Bei einer "echten" alpha-Helix ist jede Aminosäure ebenfalls mit der drittnächsten verbunden, also 1 - 4, 2 - 5, 3 - 6, und so weiter. Der Abstand zwischen zwei Windungen der Helix beträgt 0,54 nm [2, 3].
Das Interessante ist jetzt, dass Aminosäuren, die in der linearen Struktur nicht benachbart sind, also zum Beispiel Aminosäuren 1 und 4, in der Helix plötzlich Nachbarn sind.
Ob sich innerhalb eines Proteins eine alpha-Helix bildet, hängt von der genetisch bestimmten Aminosäuresequenz ab. Manche Aminosäuren eignen sich eher für die Bildung einer Helix als andere Aminosäuren. Vor allem Aminosäuren mit stark verzweigten Resten "stören" den Aufbau einer Helix. Das sind vor allem Valin, Threonin und Isoleucin, aber auch Serin, Asparaginsäure und Asparagin. Manche dieser Seitenketten können selbst H-Brücken untereinander ausbilden, was den Aufbau der Helix ebenfalls stört. Prolin passt überhaupt nicht in eine Helix hinein, denn das N-Atom von Prolin kann keine H-Brücke ausbilden [3].
Auf dieser Lexikon-Seite finden Sie viele weiterführende Einzelheiten zum Thema.
Geeignet ist diese Seite vor allem für Studierende der Medizin, Biologie, Pharmazie und Chemie sowie verwandter Naturwissenschaften.
Das beta-Faltblatt
Auch die beta-Faltblatt-Sekundärstruktur wurde von Pauling und Corey entdeckt, kurz nach der alpha-Helix. Während die Aminosäure-Kette in der alpha-Helix spiralförmig gewunden ist, liegt sie in der beta-Faltblatt-Struktur ausgestreckt vor, also als lange lineare beta-Kette:

Ein Hexapeptid als beta-Kette
Autor: Ulrich Helmich, Lizenz: Public domain
Oben sehen Sie ein Molekül-Modell einer solchen beta-Kette. Die Seitenketten werden durch grüne Kugel repräsentiert.
Die langen beta-Ketten können auf vielfältige Weise angeordnet sein. Sie können beispielsweise parallel oder antiparallel verlaufen.
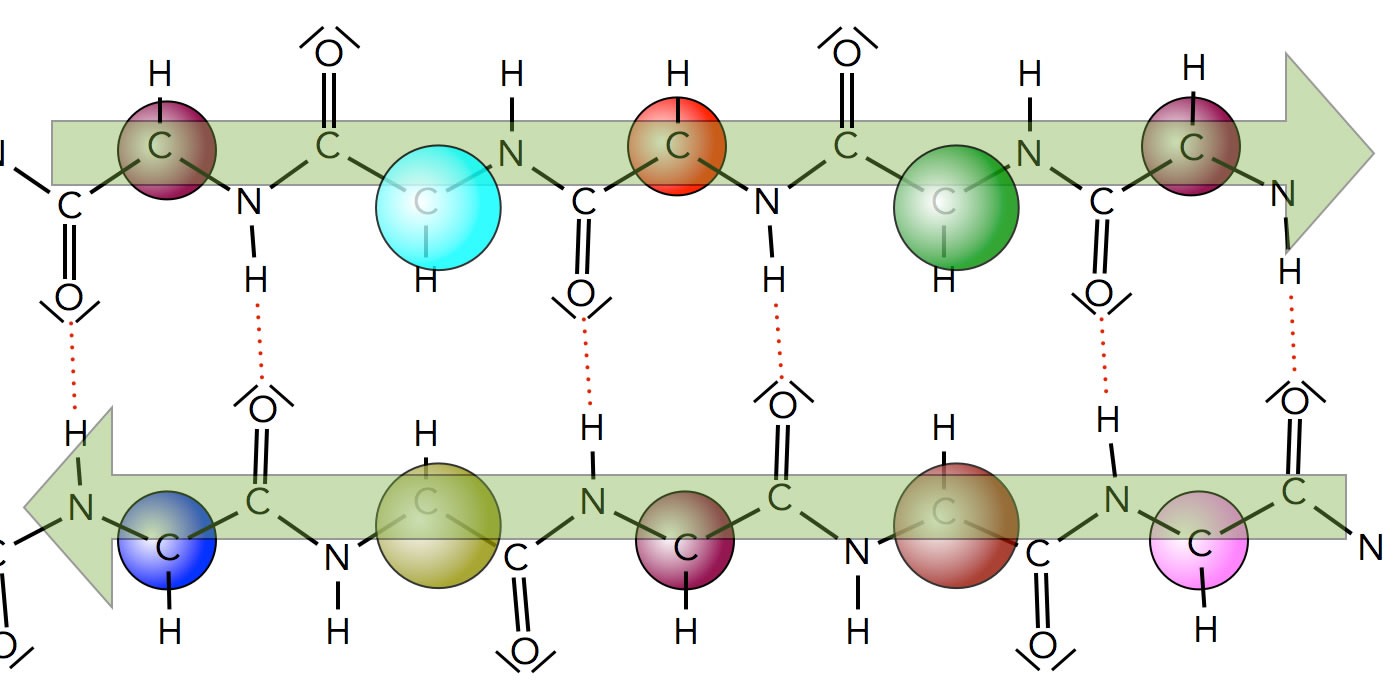
Ein antiparalleles beta-Faltblatt
Autor: Ulrich Helmich, Lizenz: Public domain
Bei dem antiparallelen beta-Faltblatt sind zwei Peptidketten in entgegengesetzt verlaufender Richtung angeordnet. Jede Aminosäure auf dem einen Strang ist mit genau einer Aminosäure auf dem anderen Strang durch H-Brücken verknüpft. Die Seitenketten (farbige Kugeln) befinden sich abwechselnd mal hinter, mal vor dem Peptid-Rückgrat.
Es gibt in vielen Proteinen auch eine parallele Anordnung der beta-Ketten. Hier verlaufen beide Ketten in der gleichen Richtung.
Meistens sind mehr als zwei beta-Ketten zu einer Faltblattstruktur verbunden.
Ein häufiges beta-Faltblatt-Motiv in Proteinen
Autor: Ulrich Helmich, Lizenz: Public domain
Hier sieht man ein Motiv, das in vielen Proteinen vorkommt, vor allem in Transmembranproteinen, die also die gesamte Zellmembran durchziehen. In der Abbildung sind die acht beta-Stränge flächig angeordnet, also in einer Ebene. Stellen Sie sich nun vor, diese Ebene würde aufgerollt zu einer Röhre. Das käme einem wirklichen Protein schon viel näher als die einfache Zeichnung in der Abbildung oben.
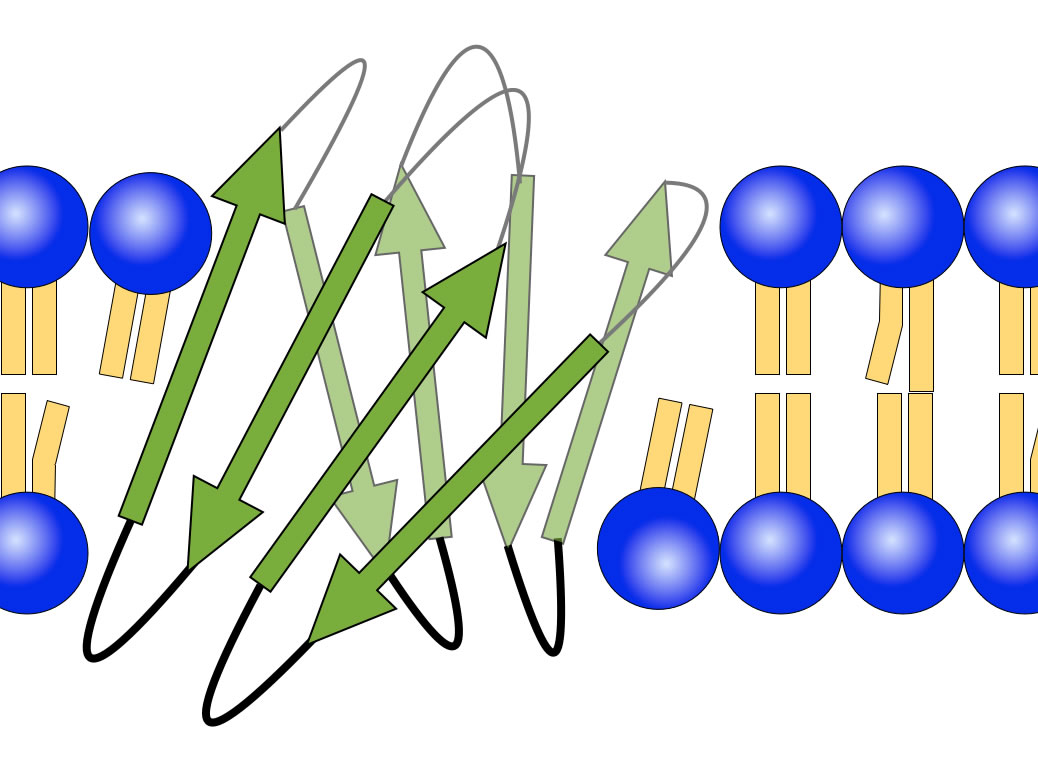
So könnten mehrere beta-Faltblätter ein Transmembranprotein in der Membran verankern
Autor: Ulrich Helmich, Lizenz: Public domain
Die obige Zeichnung ist vielleicht etwas "frei erfunden", aber so ähnlich könnte ein Motiv aus mehreren Faltblättern ein Transmembranprotein in der Membran verankern.
Auf dieser Lexikon-Seite finden Sie viele weiterführende Einzelheiten zum Thema.
Geeignet ist diese Seite vor allem für Studierende der Medizin, Biologie, Pharmazie und Chemie sowie verwandter Naturwissenschaften.
Ungeordnete Bereiche
Die eben genannten Sekundärstrukturen werden regelmäßig durch ungeordnete Bereiche zusammengehalten, die aber oft weit komplexere Aufgaben haben als einfach nur zwei geordnete Bereiche zusammen zu halten.
In diesem Lexikon-Artikel finden Sie mehr über dieses Thema.
Proteindomänen
Jane Richardson entdeckte 1981, dass es eine weitere Hierarchieebene zwischen Sekundärstrukturen und der Tertiärstruktur gibt. In vielen Proteinen tauchen immer wieder geordnete Bereiche auf, die sich aus mehreren alpha-Helices und/oder beta-Faltblättern zusammensetzen und die eine bestimmte Aufgabe innerhalb des Proteins übernehmen. Richardson nannte diese Bereiche "Domänen".
Die unheimlich große Vielfalt an Proteinen in den Zellen der Pflanzen und Tiere erklärt sich zum Teil auch mit dem sogenannten Domänen-Shuffling. Darunter versteht man die Neukombination von bewährten Domänen während der Evolution der Organismen:
"Although some bacteria have acquired domains from eukaryotic species by horizontal gene transfer, it is through the duplication, insertion, and deletion of genes that proteins principally acquire or lose domains and by which new combinations are generated. The process is termed domain shuffling. One way in which this occurs is through the shuffling of exons (again through insertion, duplication, or deletion). " [4]
Man nimmt an, das jede Domäne eines eukaryotischen Proteins durch ein Exon innerhalb des Gens codiert wird. Durch Neukombination solcher Exons (Exon shuffling) entstehen dann bei der Translation neue Proteine mit neuen Domänen-Kombinationen, die sich in der Evolution durchsetzen, wenn sie einen Selektionsvorteil bieten.
Wenn Sie mehr über die Proteindomänen lesen wollen, gehen Sie bitte auf diese Lexikon-Seite.
Quellen:
- Römpp Chemie-Lexikon, 9. Auflage 1992
- Alberts, Bruce et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- Stryer, Biochemie
- Gomperts et al. "Protein Domains and Signal Transduction" in Signal Transduction (Second Edition), 2009
Seitenanfang -
Weiter mit der Tertiärstruktur...
Weiter mit Proteindomänen...