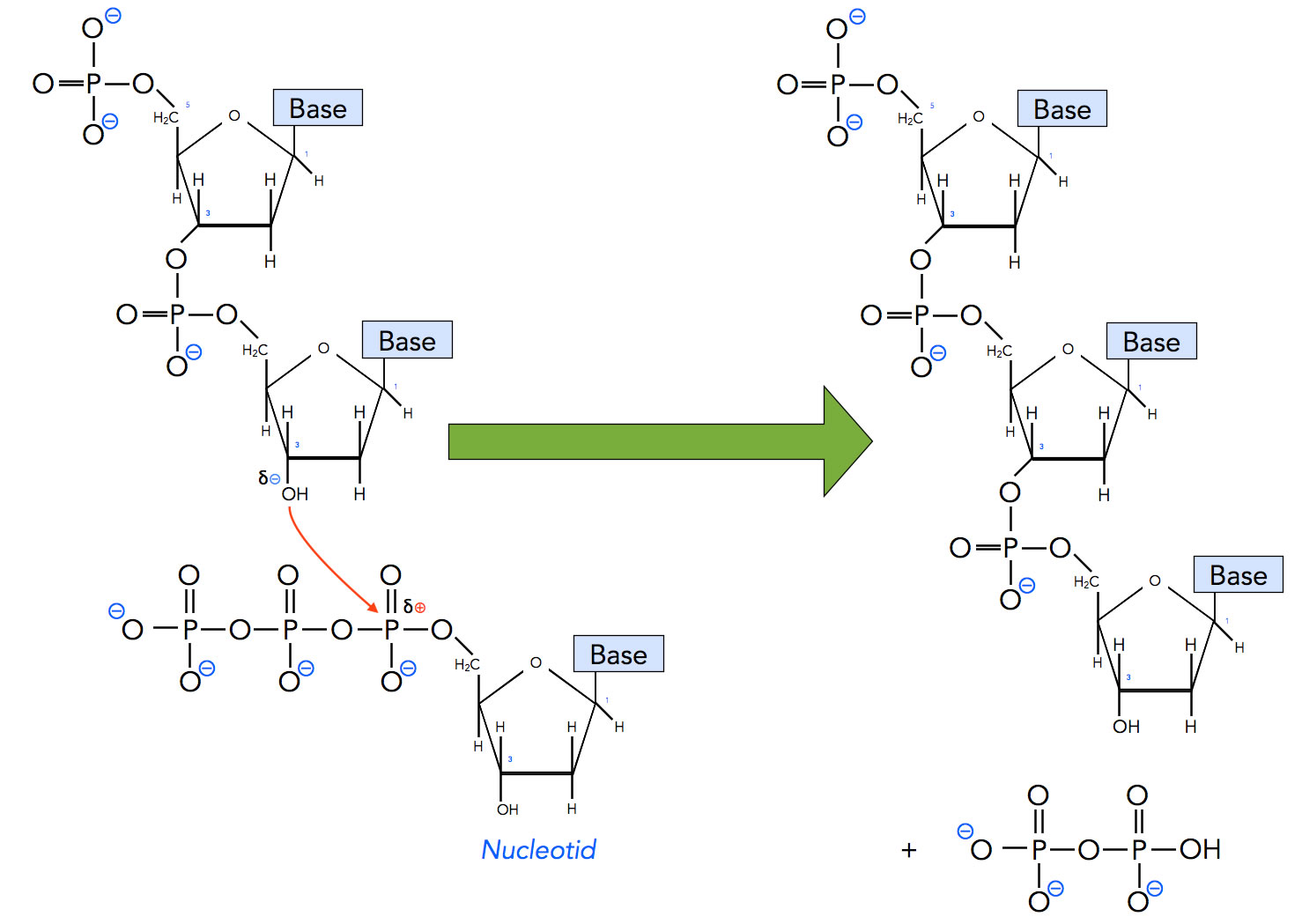
Der Elementarprozess der Elongation
Die Hauptarbeit bei der Elongation leistet die DNA-Polymerase III, ein kompliziert aufgebauter Multienzymkomplex. Wir wollen hier aber zunächst einmal die Sache vereinfachen und betrachten die Polymerase wie ein "gewöhnliches" Enzym. Was macht die DNA-Polymerase während der Elongation genau? Schauen wir uns dazu das folgende Bild an:
Links im Bild sehen wir das 3'-Ende eines DNA-Einzelstrangs sowie ein neues Nucleotid mit drei Phosphatgruppen (also ein Nucleosid-Triphosphat), das an das 3'-Ende dieses Einzelstrangs angebaut werden soll.
Das negativ polarisierte O-Atom der OH-Gruppe am 3'-C-Atom der Desoxyribose greift das positiv polarisierte Phosphor-Atom der inneren Phosphatgruppe des neuen Nucleotids nucleophil an.
Es entsteht eine neue kovalente Bindung, so dass das neue Nucleotid fest mit dem Einzelstrang verknüpft wird. Die beiden äußeren Phosphatgruppen des Nucleosid-Triphosphats werden im Laufe der Reaktion als Pyrophosphat PPi abgespalten. Das Pyrophosphat zerfällt dann in zwei Phosphatgruppen Pi, und bei diesem Zerfall wird viel Energie frei, die dann der Elongationsreaktion zu Gute kommt.
Wenn man sich diese Reaktion genau anschaut, wird auch klar, dass die DNA-Polymerase neue Nucleotide immer nur an das 3'-Ende eines Einzelstrangs anhängen kann, denn nur hier gibt es eine freie OH-Gruppe, die das zentrale Phosphor-Atom des nächsten Nucleotids angreifen kann.
Vorgänge am Leitstrang
Die Vorgänge am Leitstrang lassen sich relativ einfach beschreiben, bei dem Folgestrang wird die Sache allerdings komplizierter, darum kümmern wir uns aber später. Wir bleiben zunächst beim Leitstrang.
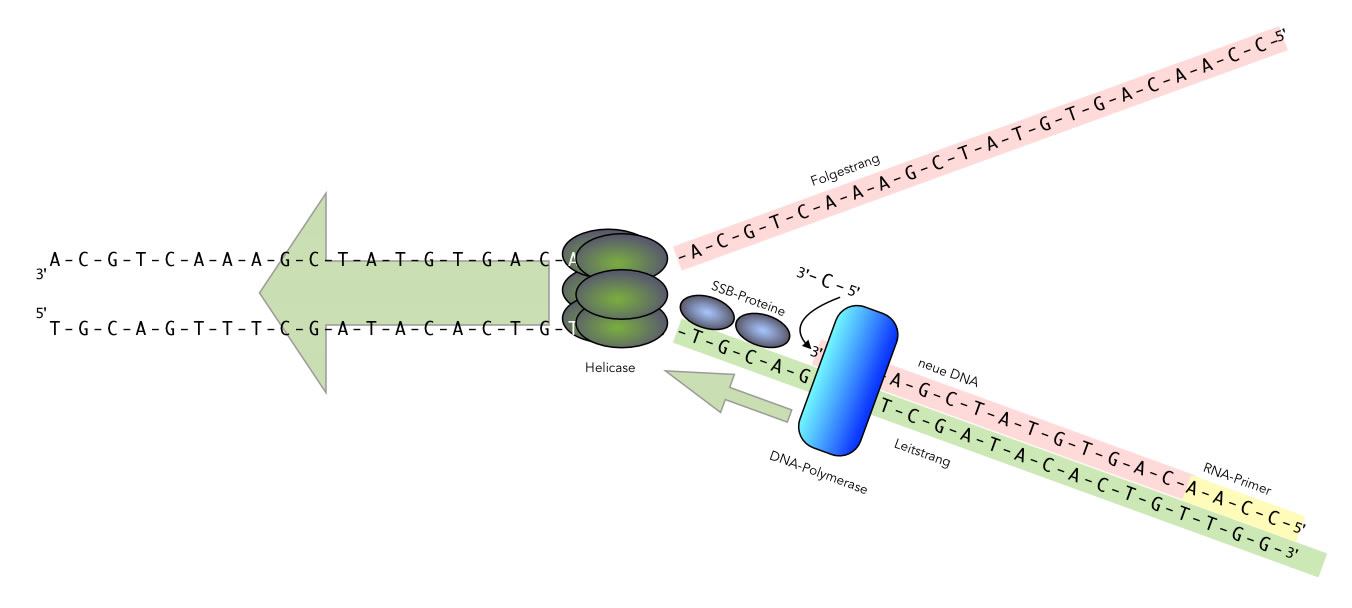
Kontinuierliche DNA-Synthese am Leitstrang
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Auf diesem Bild (stark vereinfachte Darstellung!) sieht man links den DNA-Doppelstrang und rechts die beiden Einzelstränge, die von der Helicase erzeugt worden sind. Ganz rechts auf dem Leitstrang sieht man noch einen RNA-Primer, der von der Primase während der Initiation der Replikation erzeugt wurde. Die DNA-Polymerase hat schon 13 neue Nucleotide in den wachsenden Tochterstrang eingebaut, angefangen mit C, dann A, G, T und so weiter, die beiden zuletzt eingefügten Nucleotide sieht man nicht mehr, sie werden von der Polymerase überdeckt.
Wenn Sie mehr zu diesen Enzymen der Replikation wissen möchten, gehen Sie doch einfach auf die entsprechenden Lexikon-Seiten auf dieser Homepage. Es kann natürlich sein, dass diese Seiten noch nicht ganz fertig sind, weil ich im Augenblick noch daran arbeite...
Als Nächstes soll nun ein Cytosin-Baustein in die neue DNA eingefügt werden. Dazu wird das 5'-Ende des neuen Nucleotids mit dem 3'-Ende des zuletzt eingebauten Nucleotids verknüpft, wie weiter oben ausführlich erklärt (Elementarprozess).
Die Helicase wandert nun auf der Doppelhelix weiter nach links, und die DNA-Polymerase folgt diesem Enzym auf dem einsträngigen Leitstrang und geht ihrer Arbeit nach. Die neuen Nucleotide werden immer an das 3'-Ende des wachsenden Einzelstrangs angehängt, daher sagt man, die DNA-Polymerase arbeitet in 5' → 3'-Richtung.
Die Geschwindigkeit, mit der sich die Replikationsgabel (die Baustelle sozusagen) an der DNA entlangbewegt, ist beeindruckend und leicht zu merken: ca. 50 Nucleotide pro Sekunde beim Menschen und anderen Eukaryoten, bei Bakterien und anderen Prokaryoten sogar bis zu 600 Nucleotide pro Sekunde[5].
Vorgänge am Folgestrang
Die Vorgänge am Folgestrang sind nicht ganz so einfach wie die Replikation des Leitstrangs. Ein Brite würde jetzt sagen: "Leute, wir haben ein winzig kleines Problem". So winzig ist das Problem allerdings nicht, aber Briten haben ja angeblich einen Hang zum Understatement.
Die DNA-Polymerase hängt das neue Nucleotid immer an die OH-Gruppe am C3-Atom der Desoxyribose an, also an das 3'-Ende des zu synthetisierenden neuen Einzelstrangs. Sie kann also immer nur in 5' → 3'-Richtung arbeiten. Dummerweise verläuft der Folgestrang aber genau in der entgegengesetzten Richtung (antiparallel).
Einfache Darstellung in Schulbüchern
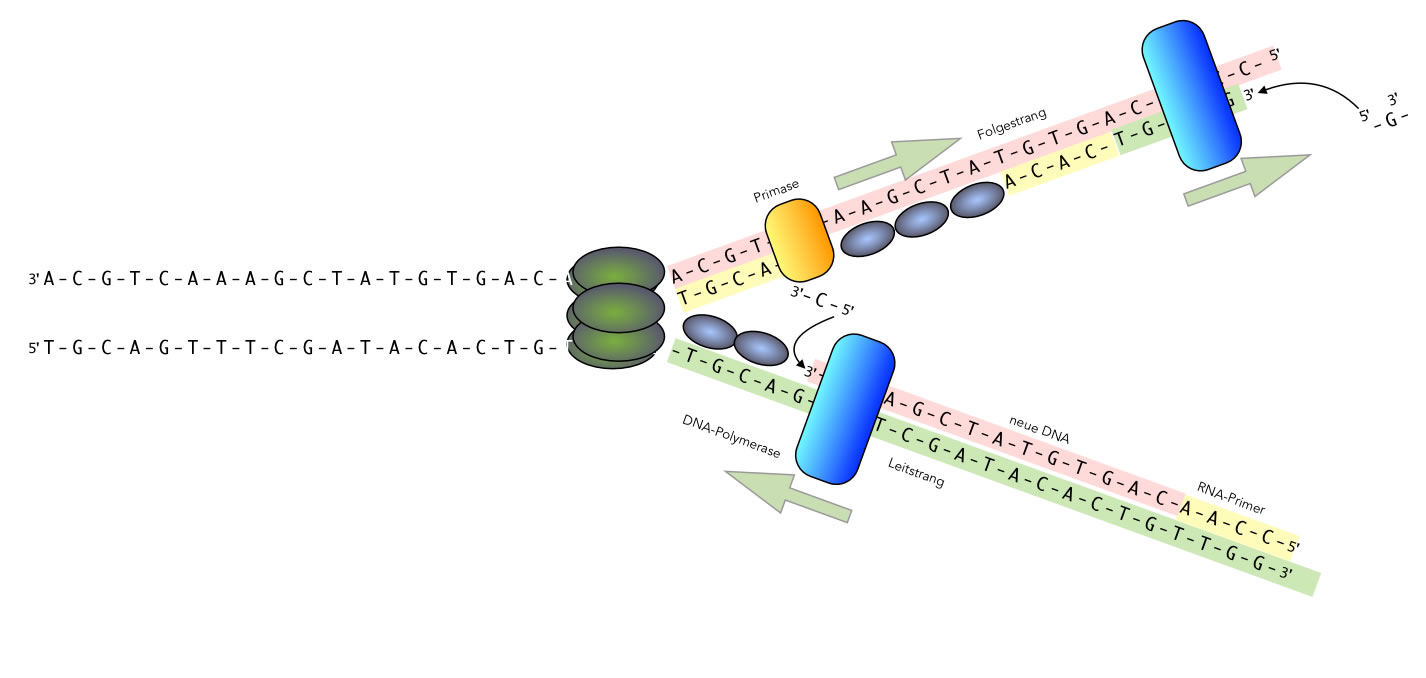
DNA-Synthese am Folgestrang
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Wir schauen uns zunächst mal eine Abbildung an, wie sie auch in vielen Schulbüchern zu finden ist. Die Vorgänge am Leitstrang haben wir bereits eben besprochen, konzentrieren wir uns jetzt auf den Folgestrang. Rechts oben im Bild sehen wir eine Polymerase, die gerade dabei ist, ein G-Nucleotid an das 3'-Ende des neu gebildeten Tochterstrangs anzubauen.
Gleichzeitig beginnt eine Primase in der Nähe der Replikationsgabel (im Bild also weiter links) einen neuen RNA-Primer zu erstellen. Zwischen diesem Primer und dem weiter rechts fertiggestellten DNA-Fragment befindet sich noch ein Einzelstrangbereich, der aber von den SSB-Proteinen stabilisiert und geschützt wird.
Wenn die DNA-Polymerase am Folgestrang mit ihrem neuen DNA-Fragment "fertig" ist, löst sie sich von der DNA und "wandert" zum 3'-Ende des gerade fertigstellten neuen Primers. Dort beginnt sie dann wieder mit der Herstellung eines Okazaki-Fragments. So heißen diese ca. 1.000 Nucleotide langen DNA-Fragmente nämlich, nach ihrem Entdecker Reiji Okazaki, der das Ganze um 1965 herausgefunden hat.
Schauen wir uns an, wie es nach einigen Millisekunden aussieht:
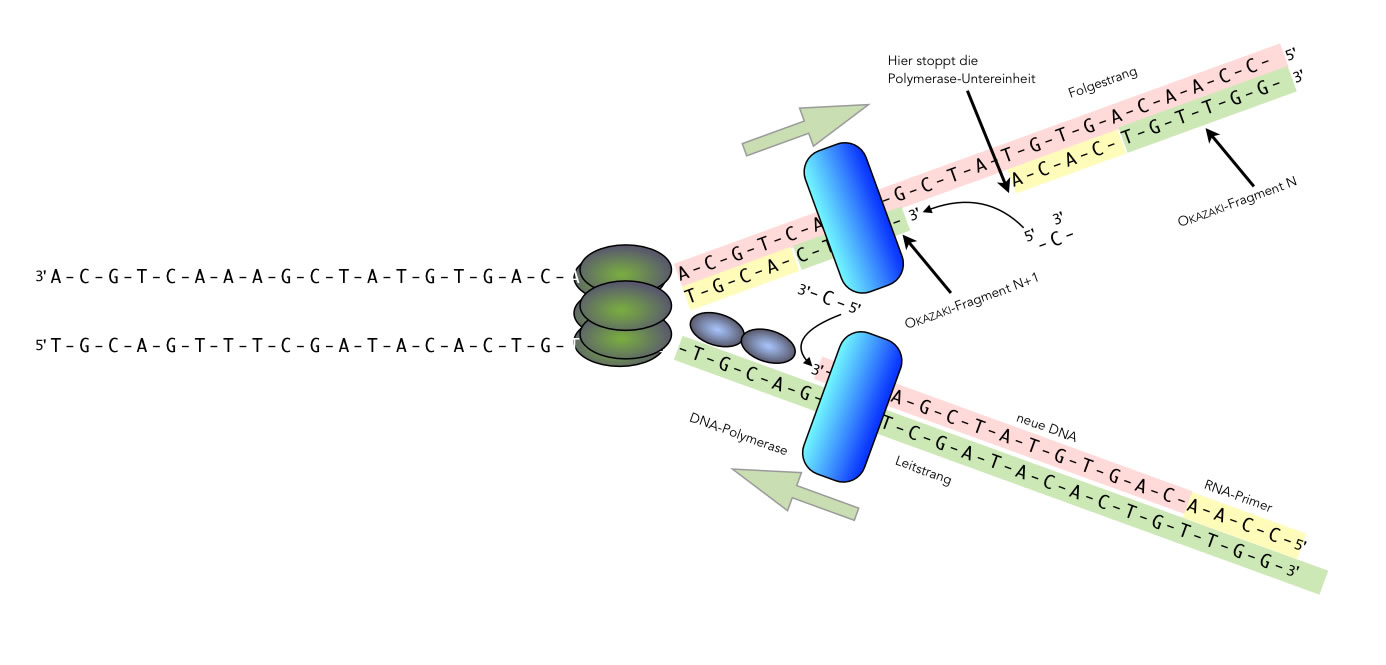
Diskontinuierliche DNA-Synthese am Folgestrang
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Die Polymerase am Folgestrang ist fertig mit "ihrem" Okazaki-Fragment (Nummer N), die Primase an der Replikationsgabel hat einen neuen Primer für Fragment (N+1) hergestellt. Die Polymerase setzt sich nun an diesen neuen Primer und beginnt wieder mit der Vervollständigung des DNA-Doppelstrangs durch Anbau neuer zum Matrixstrang komplementärer Nucleotide. Auf dem Bild sehen wir, wie gerade ein C-Nucleotid in das neue Okazaki-Fragment (Nummer N+1) eingebaut wird.
Sobald die Polymerase das 5'-Ende des rechts befindlichen Primers erreicht, stoppt die Polymerase mit ihrer Arbeit und löst sich wieder von dem Folgestrang.
Die Helicase ist inzwischen weiter nach links gewandert, es sind neue Einzelstrangbereiche entstanden, die von SSB-Proteinen stabilisiert werden, und die Polymerase auf dem Leitstrang hat weitere Nucleotide angebaut. Auf dem Folgestrang ist wieder eine Primase aktiv geworden und hat rechts neben der Replikationsgabel einen neuen Primer synthetisiert. Die DNA-Polymerase setzt wieder an dem 3'-Ende des neuen Primers an und produziert das nächste Okazaki-Fragment (N+2, hier nicht mehr eingezeichnet).
Die kurzen DNA-Stücke, die auf dem Folgestrang entstehen, werden als Okazaki-Fragmente bezeichnet. Nun gibt es aber ein weiteres "winziges" Problem, ja eigentlich sogar deren zwei:
- sind die Okazaki-Fragmente noch nicht miteinander verbunden
- befinden sich RNA-Primer zwischen den einzelnen Okazaki-Fragmenten.
Beide Probleme werden nun von einem Enzym namens Ligase beseitigt. Die Ligase "wandert" über den neu synthetisierten DNA/RNA-Strang und ersetzt die RNA-Nucleotide durch passende DNA-Nucleotide. Quasi nebenbei schließt sie dann auch noch die Lücken zwischen den "alten" DNA-Nucleotiden und den "neuen" DNA-Nucleotiden, die ja die RNA-Nucleotide ersetzt haben.
Etwas genauere Darstellung in Schulbüchern
Leider ist es aber nicht ganz so einfach. Die beiden DNA-Polymerasen sind nämlich fest miteinander verbunden und nicht frei beweglich, wie auf dem Bild angedeutet. In Wirklichkeit handelt es sich bei der DNA-Polymerase III um ein riesiges Enzym, das aus vielen Untereinheiten besteht. Zwei dieser Untereinheiten, nämlich die beiden größten, haben eine Polymerase-Aktivität. Die anderen Untereinheiten haben vielfältige andere Funktionen im Zusammenhang mit der Replikation, auf die wir hier aber nicht weiter eingehen wollen. Die Polymerase am Folgestrang kann sich also gar nicht von der Polymerase am Leitstrang entfernen, die beiden Polymerasen bleiben stets zusammen. Damit die DNA des Folgestrangs trotzdem in der korrekten Richtung verdoppelt werden kann, muss sie eine Schleife bilden und quasi "von hinten" in die Folgestrang-Polymerase eingefädelt werden.
Karteikarten-Modell der Polymerase
Vor einiger Zeit habe ich mal etwas gebastelt, um mir selbst diesen Vorgang klar zu machen. Schauen Sie sich mal folgendes Photo an:

Karteikartenmodell einer Replikation
Autor: Ulrich Helmich 2018, Lizenz: Public domain
Die beiden Papierstreifen sollen die beiden gegenläufigen Stränge der DNA-Doppelhelix andeuten. Die Pfeile zeigen die Laufrichtung der Einzelstränge an. Der linke Strang geht von unten nach oben, der rechte von oben nach unten.
Die DNA-Polymerase-Einheiten des Leit- und des Folgestrangs werden durch die beiden Schlitze in der Karteikarte dargestellt. Die Karteikarte wird von unten nach oben bewegt - in der Richtung des Leitstrangs (links). Der Folgestrang wird nun von unten in den Schlitz eingefädelt - genau wie der Leitstrang. Dazu muss der Folgestrang aber zunächst eine Schleife bilden, sonst würde er sich ja von oben in den Schlitz einfädeln. Wenn er dann aus dem Schlitz herauskommt, bewegt er sich wieder nach unten.
So ähnlich - nur viel komplexer - muss man sich die Arbeit der DNA-Polymerase bei der Replikation vorstellen. Wie das alles genau abläuft, wird in der schönen Animation gezeigt, auf die ich unten verlinkt habe.
Auf YouTube findet sich eine hervorragende (englischsprachige) Animation zu den ganzen Prozessen, die hier nur kurz angerissen wurden. Wer es sich also zutraut, sollte sich dieses schöne Video ansehen.
Noch genauere Darstellung in Schulbüchern
Wie schon oben angedeutet, findet man in manchen älteren Schulbüchern noch die einfache Darstellung mit zwei getrennten Polymerasen, einige neuere Bücher wie zum Beispiel der MARKL zeigen aber auch schon die Schleifenbildung:
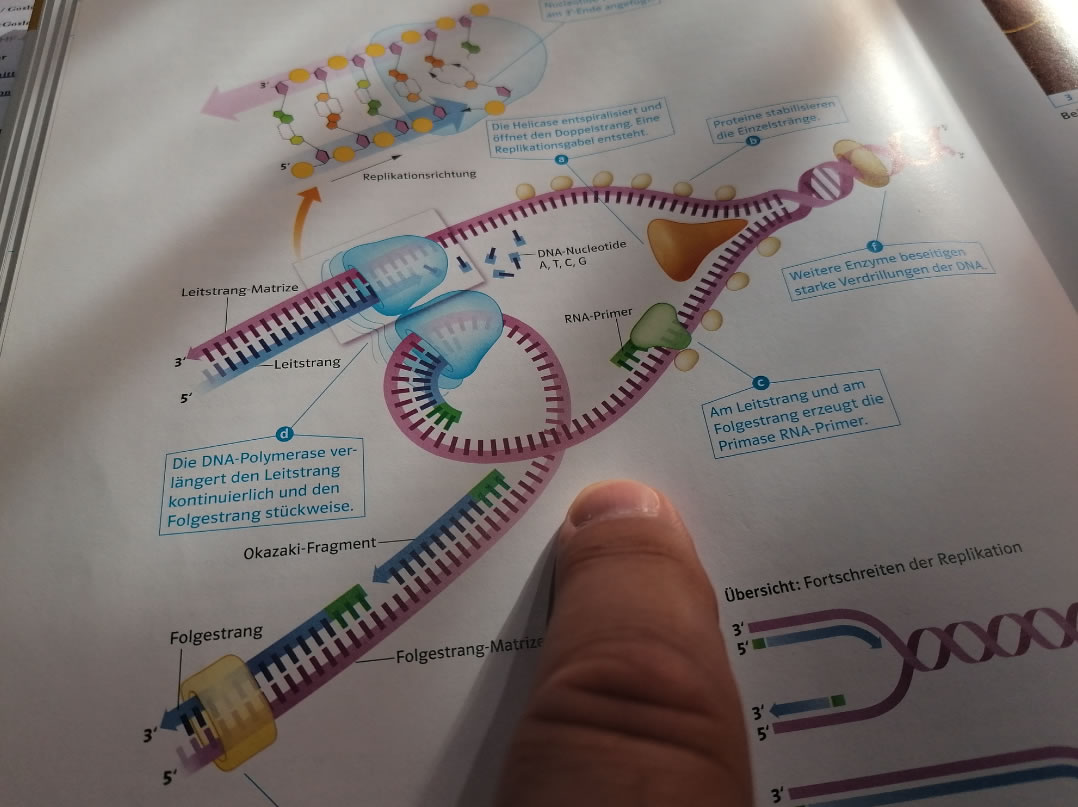
Im neuen Markl BIOLOGIE wird die Schleifenbildung schon sehr anschaulich dargestellt
Photo: Ulrich Helmich 2018.
Zwei andere renommierte Schulbücher, das Bioskop und der Linder, stellen die DNA-Replikation leider auch in den Auflagen von 2018/2019 immer noch in der vereinfachten Weise ohne Schleifenbildung dar, was aber für das NRW-Abitur völlig reicht. So weit ich weiß, wird in den schriftlichen Abiturprüfungen in NRW an keiner Stelle auf den Mechanismus der DNA-Replikation eingegangen.
Die DNA-Replikation im Zentralabitur NRW
Wird die ganze Thematik der DNA-Replikation eigentlich irgendwie im Zentralabitur berücksichtigt? Schauen wir uns doch mal die Aufgaben aus den Jahren 2007 bis 2020 an. Eine einfache Suchfunktion in den PDF-Dateien auf meiner Festplatte liefert folgende Ergebnisse zu Tage:
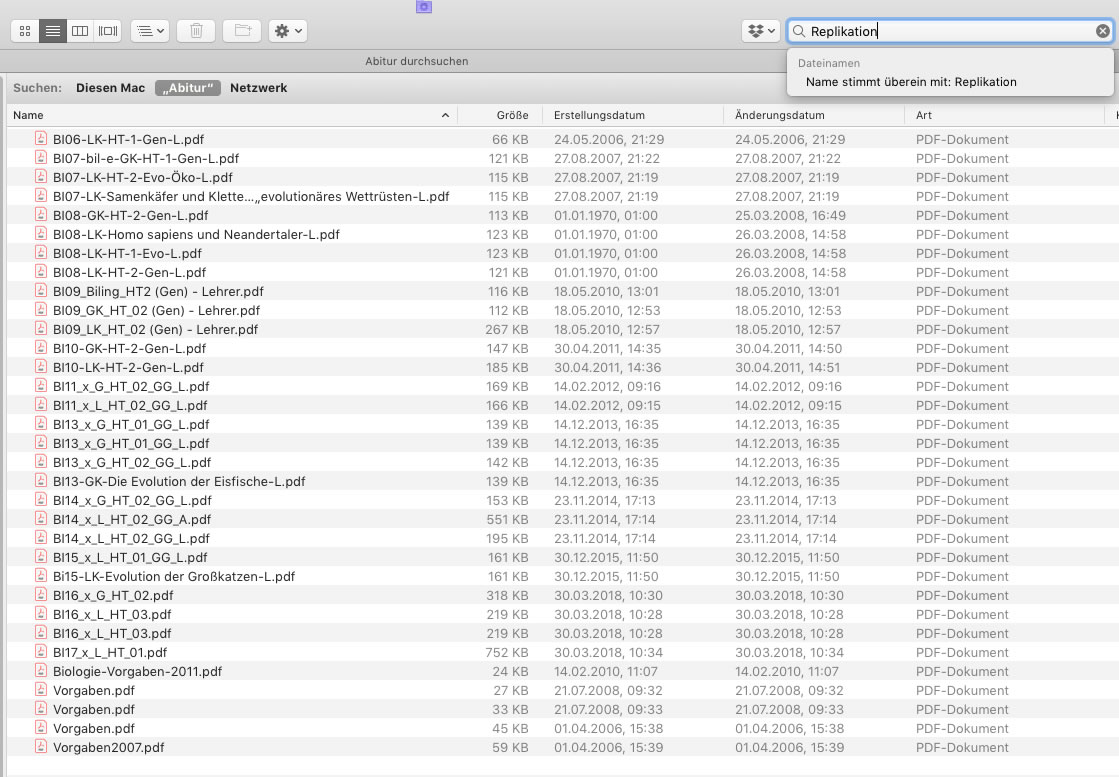
Das Stichwort "Replikation" kommt durchaus in vielen Abituraufgaben vor
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Auf den ersten Blick kommt das Stichwort "Replikation" in vielen Abituraufgaben bzw. deren Lösungen vor. Schauen wir uns eine dieser Aufgaben mal näher an, nehmen wir die aktuellste, in der das Stichwort vorkommt, die LK-Aufgabe von 2017.
Bei dieser Aufgabe, "Wirkung von Benzodiazepinen am GABAA-Rezeptor", geht es in erste Linie um Neurophysiologie. In einer Teilaufgabe wird dann auf das Verfahren der PCR eingegangen (polymerase chain reaction), bei dem ein kurzer DNA-Strang vervielfältigt wird. Die Kandidaten sollten hier aber keineswegs auf den Mechanismus der DNA-Replikation eingehen, sondern nur kurz darstellen, was PCR ist, wie diese funktioniert und welche Rolle dabei die Primer übernehmen, die man dem PCR-Ansatz zugibt: "Fassen Sie die PCR-Schritte zusammen und erläutern Sie anhand von Abbildung 3, wie die Forscher mithilfe der PCR gezielt einzelne Aminosäuren in einem Polypeptid ersetzen"
In der Abituraufgabe 3 von 2016 wird das Wort "Replikation" nur einmal am Rande erwähnt und spielt überhaupt keine Rolle bei der Aufgabenstellung, und in der Abituraufgabe 2 von 2016 - immerhin eine reine Genetik-Aufgabe ("Auswirkungen temperatursensitiver Mutationen"), wird Replikation nur in den Unterlagen für die Lehrkraft erwähnt, bei den inhaltlichen Schwerpunkten. In der Aufgabenstellung selbst oder in den formulierten Erwartungen kommt das Wort "Replikation" an keiner Stelle vor.
Im Grunde können Sie all das, was auf dieser Seite steht, für das Abitur in NRW vergessen. Da reicht es, wenn Sie wissen, dass Replikation die identische Verdopplung der DNA ist und im Zellkern abläuft. Daher richtet sich diese Seite auch eher an interessierte und engagierte GK- und LK-Schüler sowie an Studenten der Biologie und Medizin in den ersten Semestern.
Quellen:
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Nelson, Cox: LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Walter Messer: "The bacterial replication initiator DnaA". FEMS Microbiological Reviews, 1. November 2002.
Seitenanfang -
Weiter mit dem Vertiefungswissen zur Replikation...
Weiter mit der Polymerase-Kettenreaktion...