Abituraufgaben
Dieses Thema wird von folgenden NRW-Abituraufgaben behandelt:
- 2022 GK HT 3: Ivermectin-Überempfindlichkeit bei Hunden
- 2021 GK HT 3: Das Warmblood Fragile Foal Syndrome (WFFS)
- 2021 LK HT 3: Erbliche Netzhauterkrankung
Lernziele
Wenn Sie diese Seite durchgearbeitet haben, sollten Sie wissen
- wie die Polymerase-Kettenreaktion im Prinzip abläuft,
- aus welchen Phasen die PCR besteht,
- welche Anwendungsbeispiele es für die PCR gibt.
Das Grundprinzip der Polymerase-Kettenreaktion (engl. polymerase chain reaction = PCR) ist ganz einfach: "Man braucht nur ein Reagenzglas, ein paar Zutaten und eine Wärmequelle", so Kary B. Mullis, der Erfinder der PCR [3]. In den USA gibt es sogar eine Firma, die ein PCR-Kit für ca. 40 Dollar an Privatleute verkauft, damit sie in ihrer Küche mit einfachsten Hilfsmitteln ihre eigene DNA vervielfältigen können [4].
Erfindung der PCR
Das Verfahren der PCR wurde im Jahre 1983 von dem Amerikaner Kary Banks Mullis erfunden, als er nachts im Mondschein mit seiner Freundin im Auto unterwegs war. Behauptet er jedenfalls. Die Idee wurde von seinen Kollegen zunächst belächelt. So einfach, wie Mullis es darstellt, kann man doch keine DNA vervielfachen, dachten die meisten wahrscheinlich. Dann erkannten die Biologen aber langsam das gewaltige Potenzial, das hinter dieser PCR-Methode steckt. Mullis erhielt von seiner Firma die lächerlich geringe Summe von 10.000 Dollar als Anerkennung für seine Erfindung. Später verkaufte diese Firma das PCR-Patent für 3 Millionen Dollar weiter (aus heutiger Sicht auch eine sehr geringe Summe für eine solch wichtige Erfindung).
1993 erhielt Mullis dann aber den Nobelpreis für Chemie, so dass er doch noch eine angemessene Anerkennung für seine Erfindung erhielt.
Grundprinzip der PCR
1. Denaturierung
Bei 95°C wird die doppelsträngige DNA, die vervielfältigt werden soll, aufgeschmolzen (in Einzelstränge gespalten).
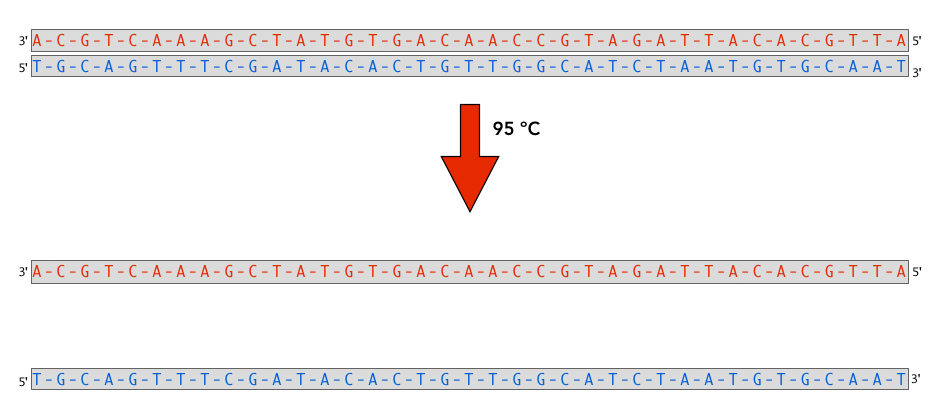
Aufschmelzen der DNA
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Die H-Brücken, welche die Basenpaare zusammenhalten, sind zwar einzeln betrachtet nur sehr schwach, in ihrer Gesamtheit jedoch entwickeln die vielen H-Brücken eines Doppelstrangs eine ganz schöne Bindungskraft. Proteine denaturieren bereits bei 40 oder 50 ºC, während die DNA erst bei über 90 ºC aufschmilzt.
2. Anlagerung der Primer
Die beiden DNA-Primer-Moleküle, die schon vorher zugesetzt worden sind, lagern sich bei 60°C an die Einzelstränge an.
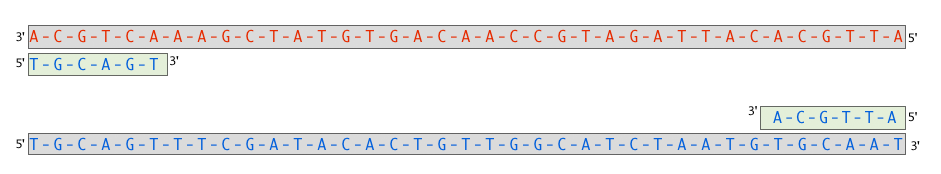
Anlagerung der beiden Primer
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Wenn die DNA aufgeschmolzen ist, liegen zwei DNA-Einzelstränge mit komplementärer Basensequenz vor. In der jeweiligen 5' → 3'-Richtung gelesen, hat aber jeder Einzelstrang eine andere Basensequenz. Es werden daher zwei verschiedene DNA-Primer benötigt, für jeden Einzelstrang muss ein eigener Primer synthetisiert werden. Dazu muss natürlich die Basensequenz zumindest der beiden Primer-Regionen vorher bekannt sein, damit man die Primer gezielt synthetisieren kann.
Die benötigten Primer werden chemisch hergestellt. Dazu muss natürlich die Basensequenz am Anfang und am Ende der zu vervielfältigenden DNA bekannt sein. Von den Primern hängt es also ab, welcher DNA-Abschnitt überhaupt kopiert und vervielfältigt wird.
3. Polymerisierung
Zwei DNA-Polymerasen synthetisieren, ausgehend von den Primern, bei 72°C die komplementären DNA-Stränge in der jeweiligen 5' → 3'-Richtung. Das heißt, an das 3'-OH-Ende des jeweiligen Primers wird das erste DNA-Nucleotid angehängt. Auf diese Weise werden aus der zu untersuchenden DNA zwei identische doppelsträngige DNA-Moleküle.
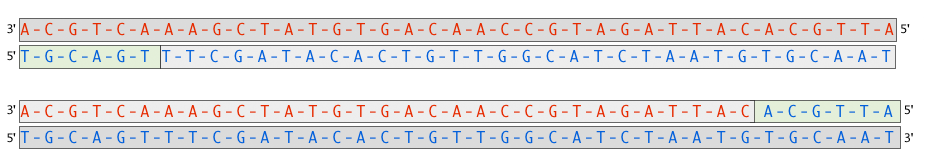
Synthese der Tochterstränge
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Für die PCR wird eine spezielle DNA-Polymerase verwendet. Eine normale Polymerase würde bereits bei einer Temperatur von 40 oder 50 ºC denaturieren, wäre also bei 72 ºC nicht zu gebrauchen. Aber es gibt ja bekanntlich Prokaryoten, die in heißen Schwefelquellen leben und locker Temperaturen von 90 oder sogar 100 ºC aushalten. Ein bekanntes Beispiel ist Thermus aquaticus. Deren DNA-Polymerasen werden jetzt als Werkzeuge für die PCR eingesetzt. Man bezeichnet diese speziellen hitzeresistenten DNA-Polymerasen auch als Taq-Polymerasen, nach dem Bakterium Thermus aquaticus.
4. Die weiteren Zyklen
Jetzt ist der erste Zyklus der PCR beendet, ein DNA-Doppelstrang wurde identisch verdoppelt. Ein solcher PCR-Zyklus dauert ca. 5 Minuten. Schauen wir uns den nächsten Zyklus noch einmal kurz an:
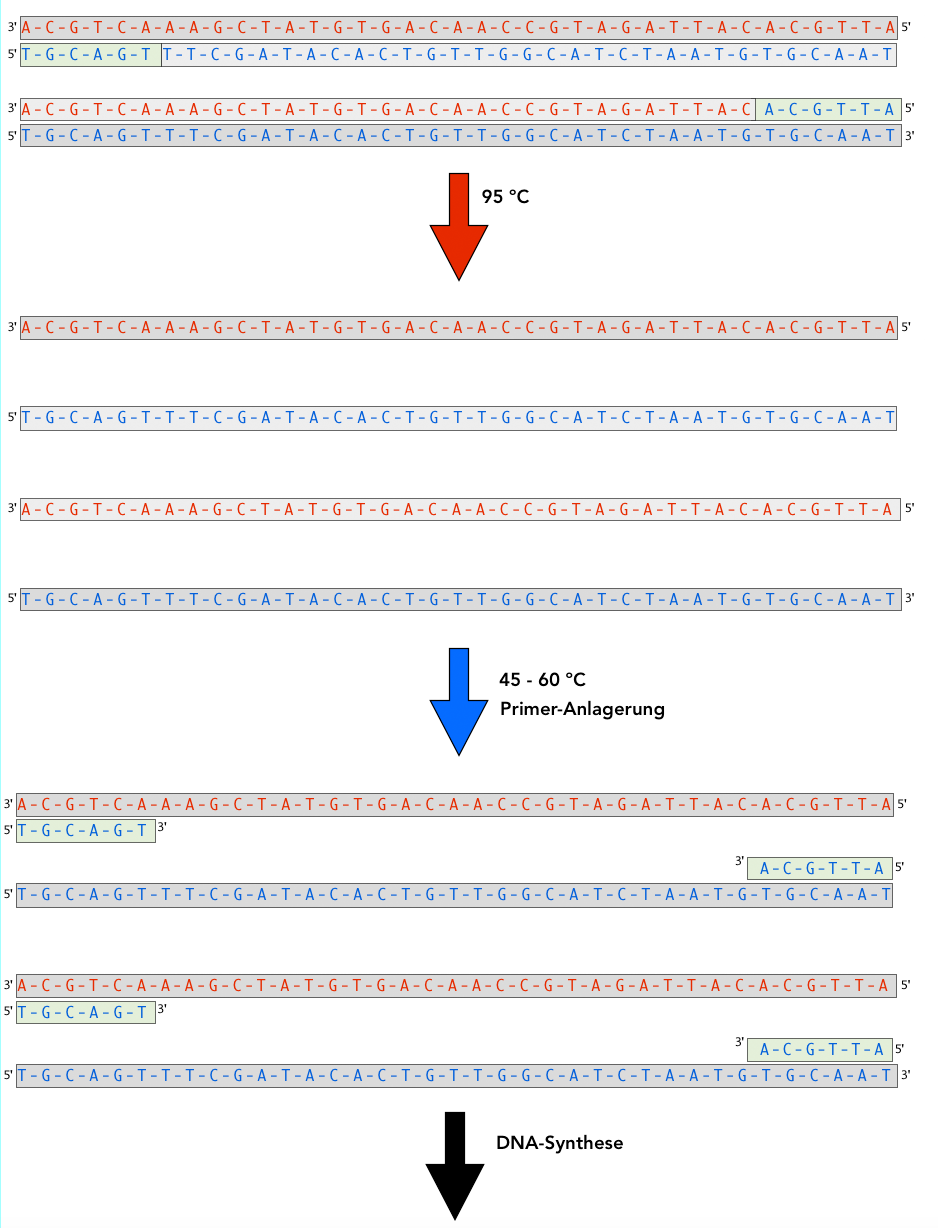
Der nächste Zyklus: Aufschmelzen der DNA, Anlagerung der Primer, DNA-Synthese (nicht mehr eingezeichnet)
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Ich denke, dass wir an dieser Stelle aufhören können. Die Taq-Polymerasen synthetisieren wieder die Tochterstränge, dann folgt wieder der Schritt 1 (Aufspalten der Doppelstränge), der Schritt 2 (Anlagerung der Primer) und der Schritt 3 (DNA-Synthese), und so geht das immer weiter.
Die Anzahl der DNA-Polymerase-Moleküle und die Zahl der eingesetzten DNA-Primer wächst natürlich mit jedem Zyklus. Am Anfang mussten nur 2 Primer für jede Ur-DNA eingesetzt werden. Nach dem 1. Zyklus sind bereits 4 Primer notwendig, nach dem 2. Zyklus 8 Primer-Moleküle und so weiter. Das Gleiche gilt für die Moleküle der DNA-Polymerase. Aber da die PCR in vitro ("im Glas") durchgeführt wird, ist die Zugabe dieser wichtigen Moleküle kein Problem.
Sie können hier eine Graphik herunterladen, die drei komplette PCR-Zyklen zeigt. Für diese Webseite ist die Graphik zu groß (vor allem zu lang), daher habe ich sie in eine eigene Datei ausgelagert.
Thermocycler
Ein ganzer Zyklus dauert nur wenige Minuten, weil man heute programmierbare Geräte einsetzt, so genannte Thermocycler, die die PCR quasi von selbst durchführen. Nach 30 solcher Zyklen hat man bereits 1 Milliarde DNA-Kopien aus der ursprünglichen DNA gefertigt. Das reicht dann auch für die meisten Zwecke.
Anwendungsgebiete der PCR
Forensik
Das bekannteste Anwendungsgebiet der PCR ist sicherlich die Erstellung eines genetischen Fingerabdrucks (genetic fingerprint). Dieses Verfahren wird in der Kriminalistik zur Erkennung von Tätern eingesetzt. Jeder Täter hinterlässt Spuren am Tatort, sei es in Form von verlorenen Haaren, Hautschuppen, Blut, Sperma oder Speichel. Ein einziges Haar genügt den Biologen und Gerichtsmedizinern, um ein genetisches Profil des Täters zu erstellen. Dazu wird die Täter-DNA zunächst vervielfacht, und zwar mithilfe der PCR.
Von den in Frage kommenden Verdächtigen werden Speichelproben genommen und ebenfalls der PCR unterworfen.
Die Täter-DNA und die Verdächtigen-DNA wird nun auf eine gelartige Folie gegeben und dann einer Gelelektrophorese unterzogen. Die Täter-DNA ergibt im UV-Licht ein typisches Bandenmuster, die DNA der Verdächtigen ebenfalls. Selbst der biologische Laie kann auf den ersten Blick erkennen, ob das Bandenmuster eines der Verdächtigen mit dem des Täters übereinstimmt.
Auf dieser Seite wird das Verfahren des genetischen Fingerabdrucks ausführlich mit Bildern erklärt.
Antigen-Nachweis
Ein anderes Anwendungsbeispiel ist die Medizin. Mit der PCR man sehr schnell fremde DNA im körpereigenen Gewebe nachweisen. So kann man zum Beispiel Bakterien- oder Virenerkrankungen (Corona!) nachweisen, lange bevor der Körper des Patienten Antikörper produziert hat. Auch Erbkrankheiten kann man mit diesem Verfahren leicht erkennen.
Lebensmittel-Analytik
In der Lebensmittelanalytik nutzt man die PCR, um fremde Gene in Lebensmitteln nachzuweisen.
Evolutionsbiologie
In der Evolutionsbiologie schließlich kann man mit Hilfe von PCR und genetischem Fingerabdruck den Verwandtschaftsgrad zwischen verschiedene Arten und Gattungen relativ genau bestimmen, so dass man bessere und genauere Stammbäume aufstellen kann.
Verwandtschafts-Analyse / Vaterschaftsnachweis
Übrigens kann man die PCR / Fingerabdruck - Methode auch zur Rekonstruktion menschlicher Stammbäume nutzen; ein Vaterschaftsnachweis ist heute kein Problem mehr.
Quellen:
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Alberts, Bruce et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- "Nobelpreis für Chemie - automatische Genvervielfältigung und gezielte Mutagenese", Spektrum der Wissenschaft 12/1993, S. 16.
- "PCR at Home", Scientific American 7/2000, S. 103.
Seitenanfang -
Weiter mit Fehlern bei der DNA-Replikation....