1. Initiation
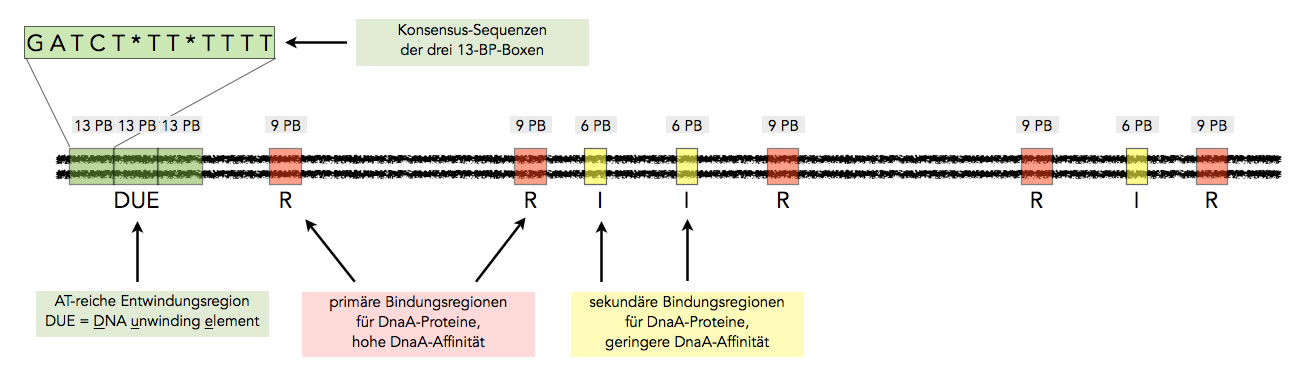
Die oriC-Region von Escherichia coli nach Lehninger [2]
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Auf diesem Bild sieht man schematisch und vereinfacht die oriC-Region des Bakteriums Escherichia coli, den Replikationsursprung. Interessant sind vor allem drei Typen von "Boxen" innerhalb dieser 245 [2] bzw. 250 [1, 5] Basenpaare (einer von den drei Autoren hat da wohl nicht richtig nachgezählt).
Die DUE-Box (DNA unwinding element) besteht aus drei Abschnitten mit je 13 Basenpaaren der Sequenz 5'-GATCT*TT*TTTT-3' [2]. Diese insgesamt 39 Basenpaare lange Box ist also reich an A=T-Basenpaaren, die relativ leicht zu spalten sind, weil sie ja nur durch je 2 H-Brücken zusammengehalten werden.
Weiterhin finden sich in oriC vier[1, 5] bzw. fünf [2] R-Boxen, die jeweils 9 Basenpaare lang sind und drei I-Boxen mit je 6 Basenpaaren [2, 3].
Erster Abschnitt der Initiation
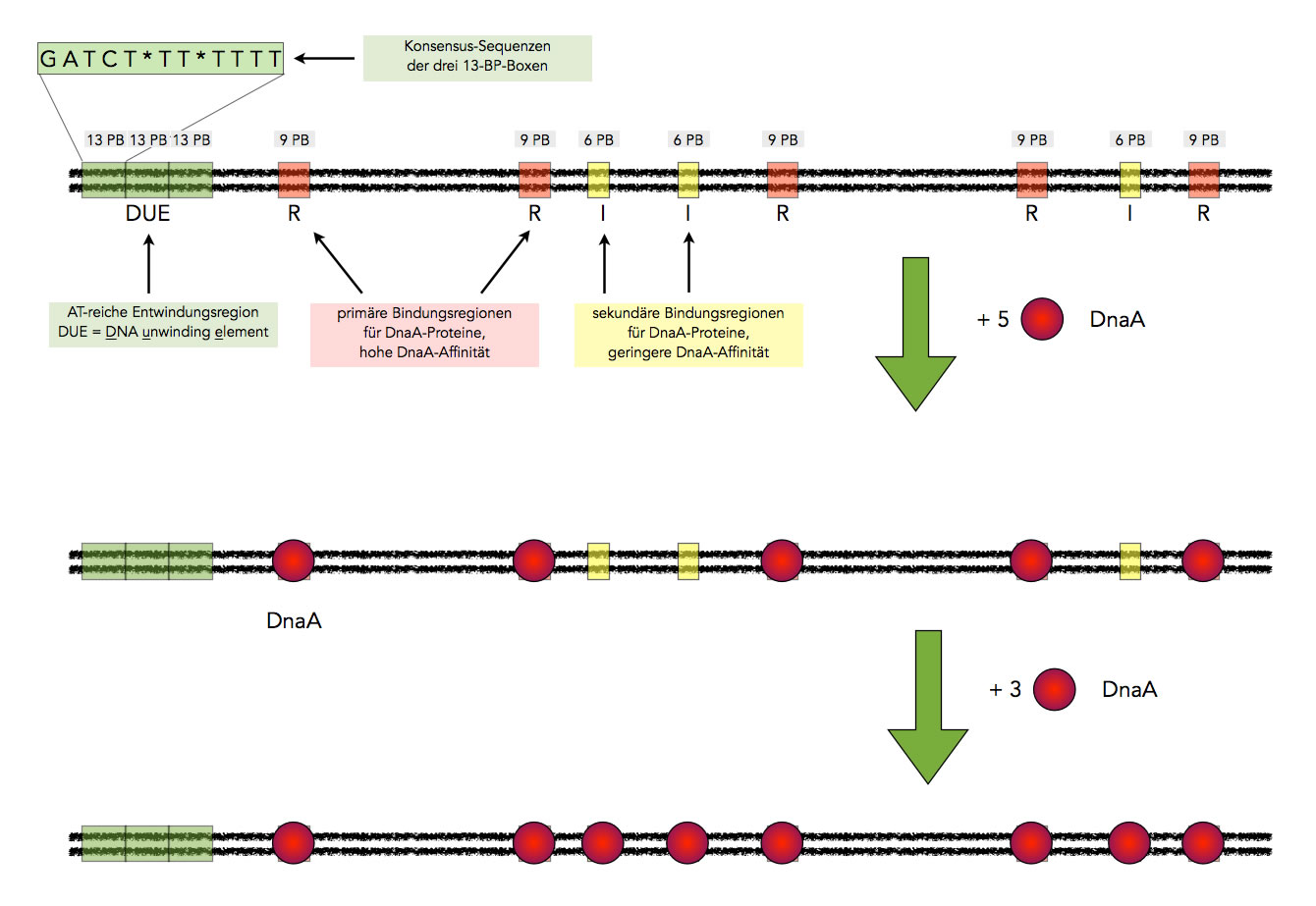
Die ersten Schritt der Initiation
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Auf diesem Bild sehen wir nun die ersten Schritt der Initiation der Replikation. Zunächst docken zunächst fünf Exemplare des Replikationsinitiationsfaktors DnaA an die R-Boxen an. Die R-Boxen haben eine sehr hohe Affinität für diese Proteine, darum werden sie zuerst besetzt. An die I-Boxen docken dann noch drei weitere DnaA-Moleküle an; die Affinität der I-Boxen für DnaA ist etwas geringer [2].
Die DnaA-Proteine müssen natürlich schon hergestellt worden sein, das geht über die normale Proteinsynthese. Das Gen für das DnaA-Protein sitzt bei den verschiedenen Bakterienarten an unterschiedlichen Stellen. Bei E. coli beispielsweise befindet es sich ca. 44.000 Basenpaare von oriC entfernt. Bei anderen Bakterienarten wie Bacillus subtilis liegt das DnaA-Gen mitten in den 2.150 Basenpaaren der oriC-Region [1].
Nach ihrer Synthese müssen die DnaA-Proteine zunächst durch ATP aktiviert werden, damit sie gut an die R- und I-Boxen binden können [1, 5].
Auf dieser Lexikon-Seite erfahren Sie mehr über das DnaA-Protein-
2. Abschnitt der Initiation
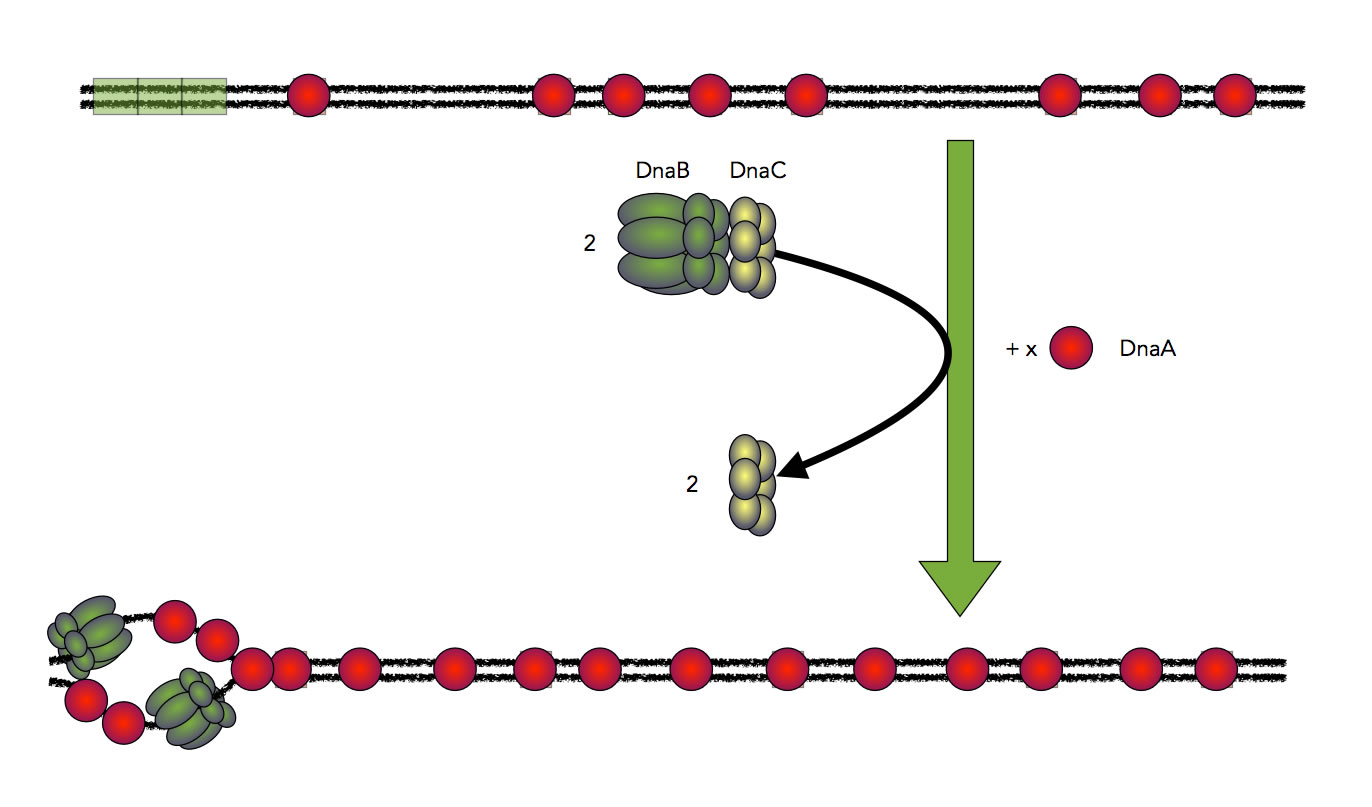
Der nächste Schritt der Initiation
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Zunächst binden weitere DnaA-Proteine an die noch freien Stellen von oriC. Wenn so um die 20 bis 30 DnaA-Einheiten zusammengekommen sind [1, 5], verbinden sich diese zu einem Proteinkomplex. Um diesen Proteinkompex wickelt sich die doppelsträngige DNA dann in Form einer Schleife [5], was durch weitere, hier nicht genannte Proteine noch unterstützt wird.
Durch diese Schleifenbildung der DNA entstehen Spannungen in der Doppelhelix, die schließlich dazu führen, dass sich die Doppelhelix in der DUE-Box öffnet; die H-Brücken zwischen den komplementären Basen lösen sich auf. Die AT-reiche DUE-Region spaltet sich in zwei kurze Einzelstränge auf. Ursache hierfür sind einerseits die Spannungen, die durch das Anlagern der vielen Proteine an den Doppelstrang entstanden sind und andererseits die Tatsache, dass die DUE-Region reich an Adenin- und Thymin-Nucleotiden ist, die ja nur durch zwei H-Brücken zusammengehalten werden [1-5].
Nun besteht natürlich die Möglichkeit, dass sich die "frisch" getrennten komplementären Basen (A-T, G-C) wieder zusammenschließen, was ja energetisch günstiger wäre. Hier kommt nun eine weitere Proteinklasse ins Spiel, nämlich die einzelstrangstabilisierenden SSB-Proteine (single strand binding).
Erst jetzt kommen zwei weitere Proteine ins Spiel, DnaB und DnaC. DnaB ist ein ringförmiges Hexamer, besteht also aus sechs Untereinheiten, die ringförmig angeordnet sind, so dass in der Mitte noch Platz für die DNA ist. DnaC, ebenfalls ein ringförmiges Hexamer, ist eine Art Hilfsprotein für das DnaB-Protein und leitet dieses zur DUE-Region [2]. In der Abbildung 3 sind diese beiden Proteine gut zu erkennen.
Das DnaB-Protein muss sich dann ringförmig um den DNA-Einzelstrang legen. Dazu muss es aber vorher geöffnet werden, da der DNA-Einzelstrang ja kein freies Ende hat, über das sich der Ring einfach schieben könnte wie über einen Finger. Dieses Schneiden ist die zweite Aufgabe des Hilfsproteins DnaC. DnaC öffnet also den DnaB-Ring und praktiziert den DnaB-Ring dann auf den Einzelstrang. Der DnaB-Ring schließt sich dann wieder. Danach hat DnaC seine Aufgabe erledigt und "kann gehen".
Das Ganze geschieht natürlich zweimal, für jeden DNA-Einzelstrang der DUE-Region einmal. Die DnaB-Proteine erfüllen nun die Funktion einer Helicase. Eine Helicase ist ein Enzym, das unter Verbrauch von ATP auf einem DNA-Einzelstrang "wandert" und dabei weitere H-Brücken zwischen den komplementären Basen auftrennt, so dass die Doppelhelix gespalten und die Einzelstrangregionen erweitert werden.
Eine nähere Beschreibung dieser wichtigen Enzyme finden Sie auf dieser Lexikon-Seite.
Die DnaB-Helicasen arbeiten sich jetzt auf den beiden Einzelsträngen in 5' → 3'-Richtung weiter, währenddessen entwindet sich die Doppelhelix. Beide Helicasen wandern in entgegengesetzte Richtungen, die beiden Replikationsgabeln entfernen sich also voneinander, die Replikationsblase weitet sich immer mehr aus.
3. Abschnitt der Initiation
Jetzt kommt wieder ein neues Enzym ins Spiel, nämlich DnaG mit der Funktion einer Primase. Die DnaG-Einheiten auf den beiden Einzelsträngen synthetisieren kurze komplementäre RNA-Stränge, sogenannte Primer.
Primer sind wichtige Enzyme, denn sie sorgen dafür, dass die DNA-Polymerase etwas "zum Greifen" hat. Die DNA-Polymerase kann ihre wichtige Arbeit nicht "einfach so" beginnen, bei Null. Sie braucht ein 3'-OH-Ende eines DNA- oder RNA-Einzelstrangs, an den sie ihre Nucleotide mit dem 5'-Phosphat-Ende anhängen kann. Diesen Gefallen tut der Polymerase die Primase, die genau einen solchen Anfangs-Strang produziert. Aber dieser Primer besteht nicht aus DNA-Nucleotiden, sondern aus RNA-Nucleotiden. Warum das so ist, müsste ich auch erst recherchieren. Sie können mir ja dabei helfen und mir schreiben, wenn Sie Erfolg hatten.
Nun setzt sich eine DNA-Polymerase III mit ihrer τ-Untereinheit (sprich "tau") an den DnaB-Ring. Die Polymerase nimmt ihre Arbeit auf und folgt dabei dem DnaB-Ring, der Helicase. Es muss aber verhindert werden, dass die noch offenen Einzelstrangregionen wieder H-Brücken ausbilden und sich zu einem Doppelstrang vereinigen. Daher kommen weitere SSB-Proteine ins Spiel. Diese legen sich an die DNA-Einzelstränge und verhindern eine Basenpaarung. Eine zweite Funktion dieser SSB-Proteine ist der Schutz der Einzelstränge vor dem Abbau durch Nucleasen, also vor Enzymen, die DNA oder RNA abbauen. SSB-Proteine decken einen Bereich von ca. 60 Basenpaaren ab.
Eine nähere Beschreibung dieses Enzyms finden Sie auf dieser Lexikon-Seite.
Quellen:
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Nelson, Cox: LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Walter Messer: "The bacterial replication initiator DnaA". FEMS Microbiological Reviews, 1. November 2002.
- Alberts, Bruce et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- Rolf Knippers: Molekulare Genetik, 11. Auflage 2018.