Die RNA-Polymerase bindet bevorzugt an Stellen auf der DNA, die vor einem Gen liegen. Eine solche Erkennungs- und Bindungsstelle nennt man Promotor. Promotoren haben zwei Aufgaben: Erstens binden sie die RNA-Polymerase mehr oder weniger fest. Zweitens stellt der Promotor den Startpunkt der Transkription dar.
Konsensus-Sequenzen
Wenn man die Nucleotid-Sequenzen von einigen hundert verschiedenen Promotoren des Bakteriums Escherichia coli vergleicht, so stellt man immer wieder fest, dass bestimmte Basensequenzen immer wieder auftauchen. Typischerweise enthalten diese Promotor-Motive häufig T- und A-Basen. Solche bei den meisten Promotoren übereinstimmend vorhandenen Basensequenzen bezeichnet man dann als Konsensus-Sequenzen (von Konsens = Übereinstimmung).
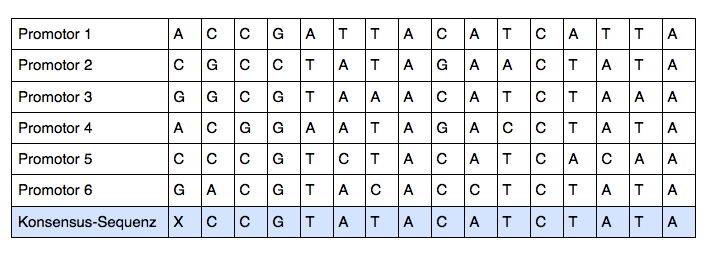
DNA-Sequenzen von sechs verschiedenen Promotoren und die daraus berechnete Konsensus-Sequenz. X steht für eine beliebige Base.
Ein Grund für das gehäufte Vorkommen der Basen T und A ist sicherlich, dass sich zwischen T- und A-Basen nur zwei Wasserstoffbrücken ausbilden. T/A-reiche DNA-Regionen können also besonders leicht aufgetrennt werden, und das ist ja auch der erste wichtige Schritt bei der Transkription der DNA. Am Promotor kann die DNA-Doppelhelix also wegen der vielen T- und A-Basen besonders leicht in Einzelstränge gespalten werden (Spektrum der Wissenschaft, März 2014, Seite 20).
Starke und schwache Promotoren
Die Konsensus-Sequenz von Promotoren ist das Ergebnis einer statistischen Analyse, so ähnlich wie in Abbildung 1 demonstriert, nur viel komplizierter. Dass irgendein Promoter genau diese Sequenz besitzt, ist zwar nicht unmöglich, aber sehr unwahrscheinlich. Jede Promotor-Sequenz wird mehr oder weniger stark von der Konsensus-Sequenz abweichen.
Es gibt aber Promotoren, deren Basensequenzen der Konsensus-Sequenz ziemlich ähnlich sind. Da sich die RNA-Polymerase nach einem abgeschwächten Schlüssel-Schloss-Prinzip an die Promotor-DNA heftet, ist es klar, dass Promotoren, deren DNA-Sequenz der Konsensus-Sequenz nahe kommt, die Polymerase besonders stark binden. Promotoren, deren DNA-Sequenz stärker von der Konsensus-Sequenz abweicht, binden die RNA-Polymerase nicht so stark. Dementsprechend unterscheidet man zwischen starken und schwachen Promotoren.
Ein typischer bakterieller Promotor
Betrachten wir dazu folgendes Bild, es zeigt den Aufbau des Promotors eines E. coli - Gens für ribosomale RNA [1, 4] (rrnB P1).
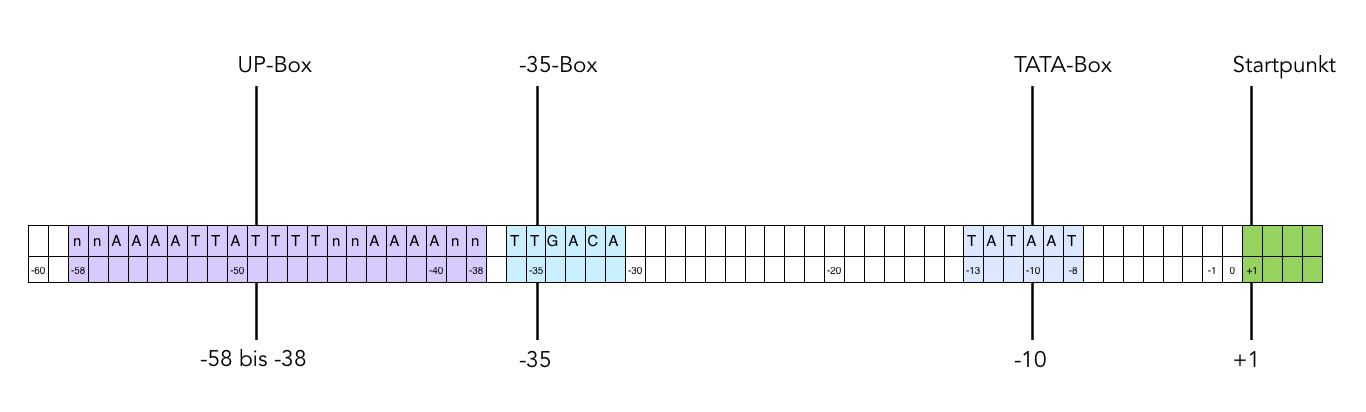
Aufbau eines typischen bakteriellen Promotors
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Drei Regionen sind hier von Interesse. Zunächst einmal die sogenannte TATA-Box, eine A- und T-reiche kurze Sequenz rund um Position -10. Die Positionsangaben beziehen sich auf den Start des eigentlichen Gens, diesem Startpunkt wird generell die Position +1 zugeteilt. Eine zweite charakteristische A- und T-reiche Sequenz befindet sich an Position -35. Schließlich gibt es noch eine relativ lange Sequenz zwischen Position -58 und -38, also direkt vor der -35-Box. Das kleine n steht hier für beliebige Basen. Bei den hier genannten Sequenzen handelt es sich stets um Konsensus-Sequenzen (siehe oben).
Die Aufgabe dieser Sequenzen ist eine doppelte:
- Erkennung des Promotors durch die RNA-Polymerase
- Bindung der RNA-Polymerase an den Promotor
Um die Bedeutung dieser drei Regionen für den Start der Transkription (Initiation) zu beschreiben, muss man zunächst einmal den Aufbau der bakteriellen RNA-Polymerase kennen.
Auf dieser Seite wird der Aufbau der bakteriellen RNA-Polymerase näher beschrieben. Sie sollten sich diese Seite kurz anschauen.
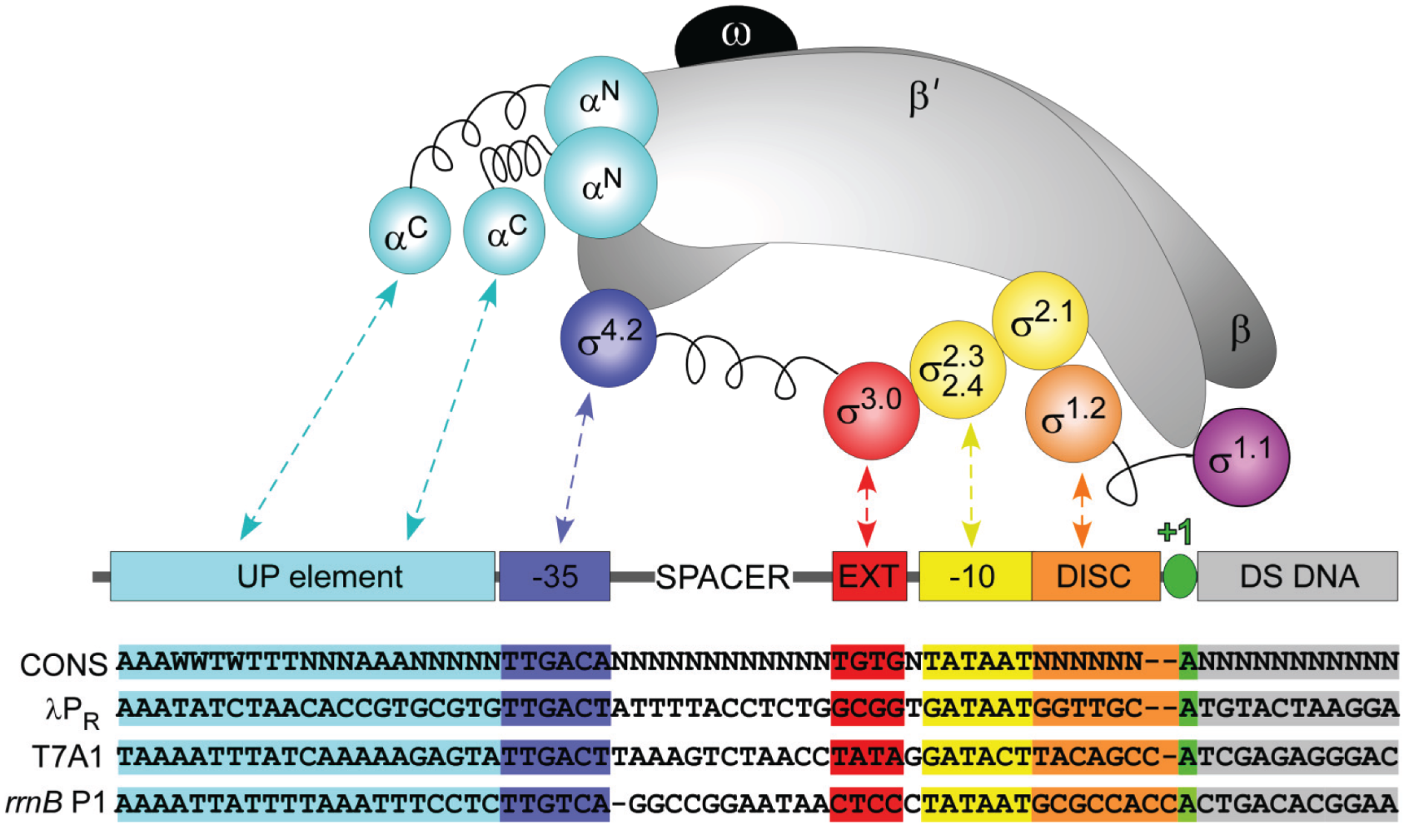
Interaktion der Untereinheiten der RNA-Polymerase mit verschiedenen Promotoren
Quelle: "Initial Events in Bacterial Transcription Initiation" [5], Autoren: Ruff, Record, Artsimovitch 2015, Lizenz: CC BY 4.0
Auf diesem Bild sieht man gut, wie die einzelnen Untereinheiten der RNA-Polymerase mit den verschiedenen Regionen des Promotors interagieren. Die sigma-Untereinheiten binden an die TATA-Box an Position -10, eine sigma-Untereinheit bindet an die -35-Box, und die C-terminalen Enden der beiden alpha-Untereinheiten binden an das UP-Element vor der -35-Box. Ein sehr ähnliches Bild findet sich übrigens in [2] auf S. 79, das ich hier aber aus verständlichen Gründen nicht zeigen kann.
Quellen:
- Alfred Nordheim, Rolf Knippers: Molekulare Genetik, 11. Auflage, Thieme-Verlag Stuttgart 2018.
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- engl. Wikipedia, Artikel "Consensus sequence".
- Estrem, Gaal, Ross and Gourse: "Identification of an UP element consensus sequence for bacterial promoters", Proc Natl Acad Sci U S A. 1998 Aug 18; 95(17): 9761–9766.
- Ruff, Record, Artsimovitch: "Initial Events in Bacterial Transcription Initiation" auf www.mdpi.com.