Einfache periphere Membranproteine
Diese Proteine liegen der Membran locker auf. Sie sind mit der äußeren oder der inneren Lipidschicht durch schwache chemische Bindungen verbunden und können sehr leicht wieder abgespült werden, zum Beispiel durch Änderungen des pH-Wertes oder Zugabe eines Salzes. Solche locker auf der Membran liegenden Proteine werden manchmal auch als angelagerte Proteine bezeichnet.
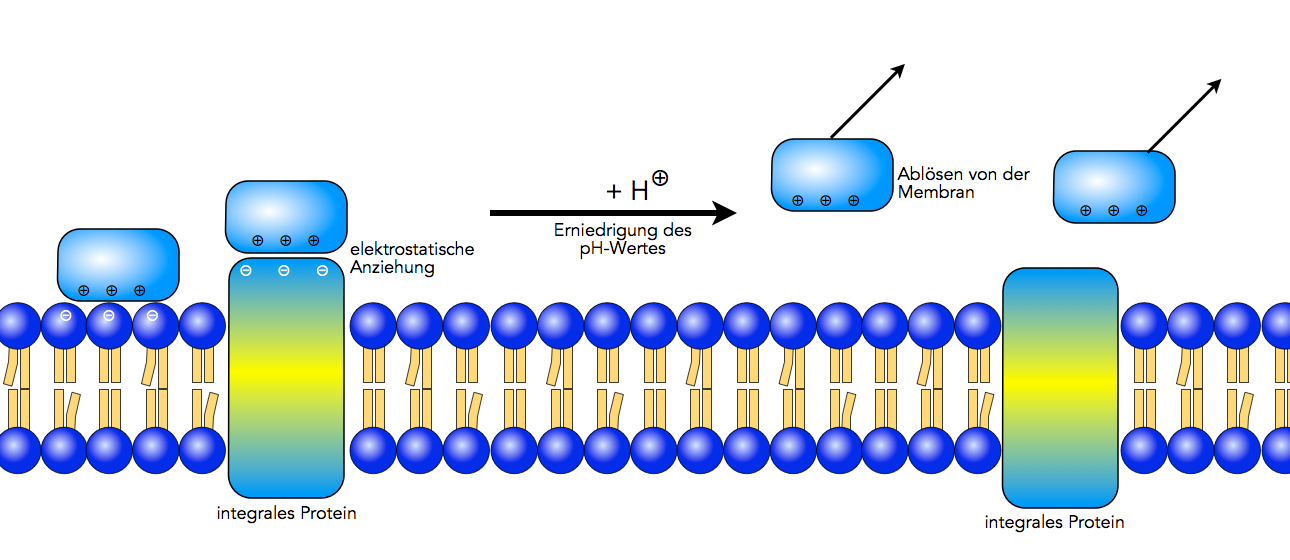
Ablösung peripherer Proteine von der Membran
Autor: Ulrich Helmich, Lizenz: Public domain
Wie die obige Abbildung zeigt, kann ein peripheres Protein durch elektrostatische Anziehungen mit einem anderen Membranprotein verbunden sein oder auch mit den Kopfregionen von Membranlipiden.
Beim Lecithin-Molekül hatten wir ja gesehen, dass in der Kopfregion elektrische Ladungen vorhanden sind. Das eine Sauerstoff-Atom des Phosphorsäure-Restes ist negativ geladen, und das Stickstoff-Atom des Cholin-Restes positiv. Die sauren und basischen Aminosäuren von Proteinen können ebenfalls elektrische Ladungen tragen: Saure Aminosäuren können ein Proton abgeben und erhalten dann eine negativ geladene Seitengruppe, und basische Aminosäuren können ein Proton aufnehmen, wodurch sie eine positiv geladene Seitengruppe erhalten.
Die Abbildung 2 zeigt, wie leicht man periphere Proteine von der Membran abspalten kann. Wird das periphere Protein durch negative Ladungen angezogen, reicht oft schon eine Erniedrigung des pH-Wertes (= Zufuhr von Protonen) aus, um das Protein von der Membran zu lösen. Die negativen Ladungen (meistens negativ geladene Sauerstoff-Atome) verbinden sich dann nämlich mit den Protonen, wobei die negativen Ladungen verschwinden und die positiven Ladungen des peripheren Proteins durch nichts mehr festgehalten werden. Das abgelöste Protein löst sich dann gut in dem wässrigen Außenmedium.
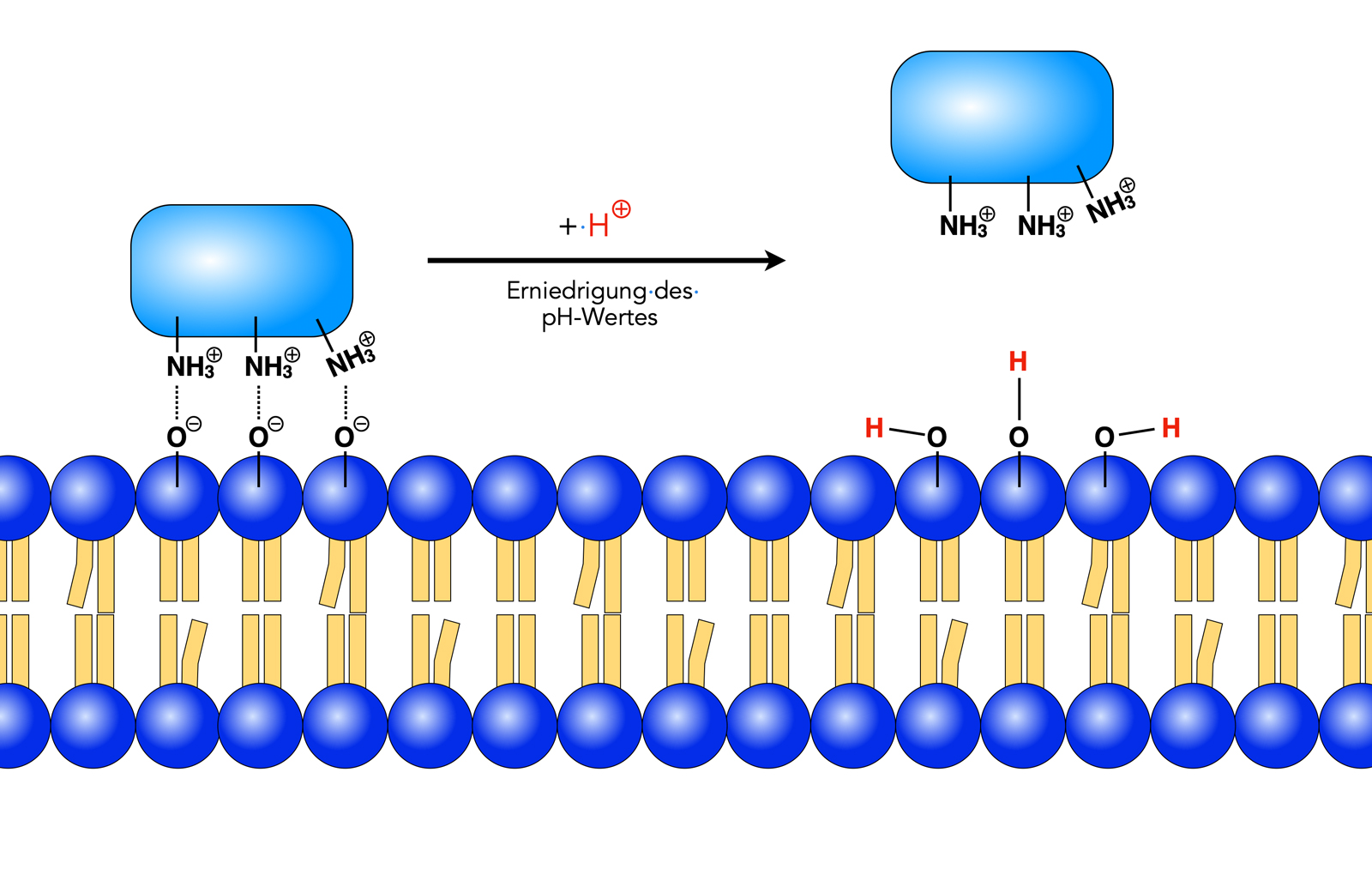
Ablösung peripherer Proteine von der Membran: Detail
Autor: Ulrich Helmich, Lizenz: Public domain
Hier sehen wir das Gesagte noch einmal im Detail veranschaulicht. Negativ geladene O-Atome in den Phosphorsäure-Resten eines Lipids bilden elektrostatische Bindungen aus zu den positiv geladenen N-Atomen in den Seitenketten basischer Aminosäuren des peripheren Proteins. Einige der zugeführten Protonen setzen sich an die negativ geladenen O-Atome, es bilden sich OH-Gruppen, und die Negativladung verschwindet. Die positiv geladenen N-Atome werden durch nichts mehr festgehalten, das Protein löst sich von der Membran.
Nicht nur Protonen können einen Einfluss auf diese Bindungen einnehmen, sondern auch Salz-Ionen, wie man am Beispiel des Cytochrom C sehen kann. Cytochrom C der inneren Mitochondrienmembran ist eines der am besten untersuchten peripheren Membranproteine. Es wird durch das integrale Membranprotein Cytochromoxidase auf der Außenseite der inneren Mitochondrienmembran verankert. Bereits durch nur 0,15 molare Kaliumchlorid-Lösung kann das Cytochrom C von der Membran getrennt werden [1]. Die Chlorid-Ionen sind negativ geladen und könnten sich theoretisch an positiv geladene N-Atome setzen, oder umgekehrt könnten sich die positiv geladenen K+-Ionen der KCl-Lösung an negativ geladene O-Atome binden.
Amphitrope periphere Proteine
Einige periphere Membranproteine werden als amphitrope Proteine (engl.: amphitropic proteins) bezeichnet. "Normale" periphere Membranproteine sind durch schwache chemische Bindungen sehr locker und vor allem reversibel mit der Membran verbunden (siehe Abschnitt oben). Durch Änderungen des pH-Wertes oder der Konzentration bestimmter Salze lösen sich diese Proteine von der Membran und diffundieren dann frei im Zellplasma oder im Außenmedium herum. Ihre Konformation ändern diese Proteine jedoch nicht, wenn sie sich von der Membran lösen oder umgekehrt an diese andocken.
Bei den amphitropen peripheren Proteinen ist das anders. Sie sind zwar auch nur sehr locker an die Membran gebunden und gehören damit zu den peripheren Proteinen. Aber das Ablösen von der Membran bzw. das Andocken an die Membran kann erstens reguliert werden und ist zweitens mit einer Änderung der Konformation verbunden: Es gibt dann eine aktive und eine inaktive Konformation. Meistens sind diese Proteine in "gelöster" Form (also wenn sie sich frei im Cytoplasma bewegen) inaktiv. Durch bestimmte Effektoren wie zum Beispiel Ca2+ können diese Proteine jedoch dazu gebracht werden, an die Membran anzudocken und gleichzeitig in eine aktive Konformation zu wechseln.
Was man jetzt eigentlich unter amphitropen Proteinen versteht, ist in der Fachliteratur noch nicht ganz endgültig geklärt. Manche Autoren fassen periphere und amphitrope Proteine zu einer Klasse zusammen, andere Autoren zählen zu den amphitrope Proteinen nur solche, die mit Lipiden kovalent verbunden sind. Ein richtig einheitliches Bild gibt es noch nicht, dazu ist die Klasse der amphitrope Proteine noch recht neu.
Nach Lehninger [2] sind die amphitropen Proteine kovalent mit bestimmten Membranlipiden verbunden. Kovalente Bindungen sind ja nicht in Stein gemeißelt, sondern können durch bestimmte Enzyme leicht wieder gelöst werden. Daher können kovalent gebundene amphitrope Proteine leicht wieder von der Membran getrennt werden.
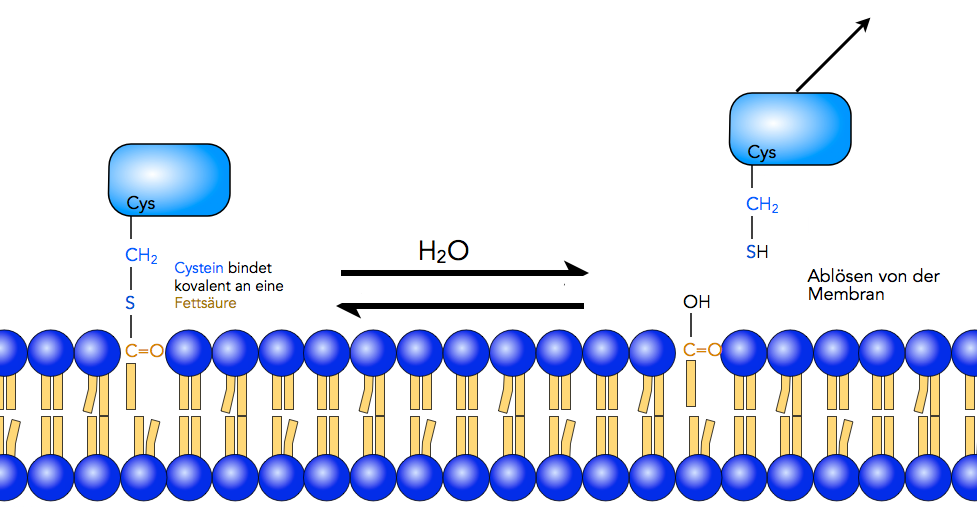
Ablösung eines amphitropen Proteins von der Membran
Autor: Ulrich Helmich, Lizenz: Public domain
Das Bild zeigt eine von mehreren Möglichkeiten, wie ein Protein kovalent aber reversibel an die Membran gebunden sein kann.
Die Aminosäure Cystein in dem Protein hat eine S-H-Gruppe in der Seitenkette, und eine freie Fettsäure hat eine COOH-Gruppe an ihrem polaren Ende. Diese beiden funktionellen Gruppen können nun unter Kondensation (Abspaltung von Wasser) eine feste Bindung eingehen (man spricht hier von einem Thioester, analog zu einem "normalen" Ester, allerdings mit einer SH-Gruppe statt einer OH-Gruppe). Das amphitrope Protein ist dann fest an die Membran gebunden. Andererseits kann diese Thioester-Bindung aber auch leicht wieder durch Hydrolyse (Zufuhr von Wasser) gespalten werden.
Na ja, ganz so einfach ist die reversible Ablösung des Proteins von der Membran auch wieder nicht. Das Protein muss dazu nämlich seine Konformation ändern, und das ist oft ein aktiver Vorgang, der eine Phosphorylierung (Anlagerung einer Phosphatgruppe), Acylierung (Anlagerung einer CH3-COOH-Gruppe) oder die Anlagerung eines Liganden benötigt.
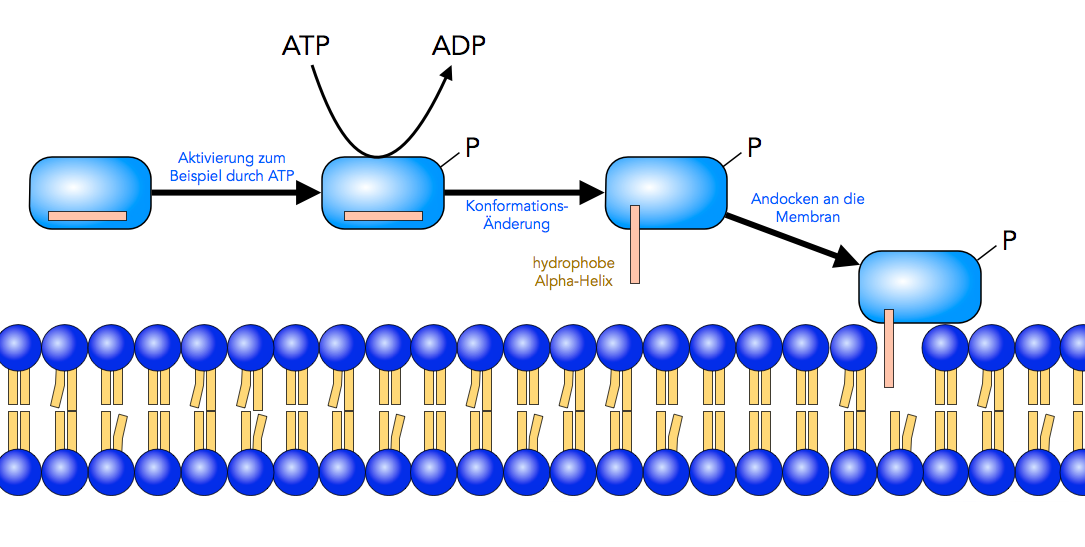
So könnte man sich vielleicht die Aktivierung eines amphitropen Proteins durch ATP vorstellen
Autor: Ulrich Helmich, Lizenz: Public domain
Diese Aktivierung und Konformationsänderung führt dann bei vielen Proteinen dazu, dass eine hydrophobe alpha-Helix, die bisher im Innern des Proteins verborgen war, plötzlich "ausgestülpt" wird, so dass sie sich in den hydrophoben Teil der Membran integrieren kann [1].
Beispiele
Zu den amphitropen peripheren Proteinen gehört eine ganze Reihe bekannter Enzyme wie zum Beispiel Cytochrom C, das eine wichtige Rolle bei der Atmungskette an der inneren Mitochondrienmembran spielt, oder Strukturproteine wie die Ankyrine. Ankyrine sind Proteine, die Fasern des Cytoskeletts mit anderen Membranproteinen verknüpfen. So verankern sie (daher auch der Name!) beispielsweise Ionenkanäle in der Zellmembran.
Auch die Phospholipasen gehören zu den peripheren Proteinen. Die Phospholipase C beispielsweise hydrolysiert das Membranlipid Phosphatidylinositol-4,5-bisphosphat (PIP2) zu zu Inositoltrisphosphat (IP3) und Diacylglycerol (DAG), zwei wichtigen second messengern, die andere Enzyme aktivieren (oder hemmen) können.
Weitere Informationen zu diesem wichtigen Enzym gibt es auf dieser Lexikon-Seite.
Die Phospholipase A2 ist ein Enzym, das in Phosphoglyceriden die zweite Fettsäure vom Glycerin abspaltet (Hydrolyse der Esterbindung am C2-Atom, daher der Namenszusatz "A2"). Oft wird dabei die Fettsäure Arachidonsäure freigesetzt. Arachidonsäure selbst ist entzündungsfördernd, gleichzeitig ist sie aber auch die Vorstufe für die entzündungshemmenden Prostaglandine.
Auch das Myelin-basische Protein (MBP) gehört zu dieser Klasse von Proteinen. Dieses Protein trägt insgesamt 20 positive Ladungen an seinen Arginin- und Lysin-Seitenketten [7] und nimmt Kontakt mit 18 Phospholipid-Molekülen auf [5].
Lipid-verankerte periphere Proteine
Als Lipid-Anker bezeichnet man bestimmte Fettsäuren (Myristinsäure- oder Palmitinsäure-Reste) oder Isopren-Oligomere (Farnesyl-, Gernaylgeranyl-Reste), die kovalent an ein Protein auf der Innenseite der Membran gebunden sind (meistens über die Seitenkette der Aminosäure Cystein) und dieses Protein in der hydrophoben Mittelschicht der Zellmembran verankern.
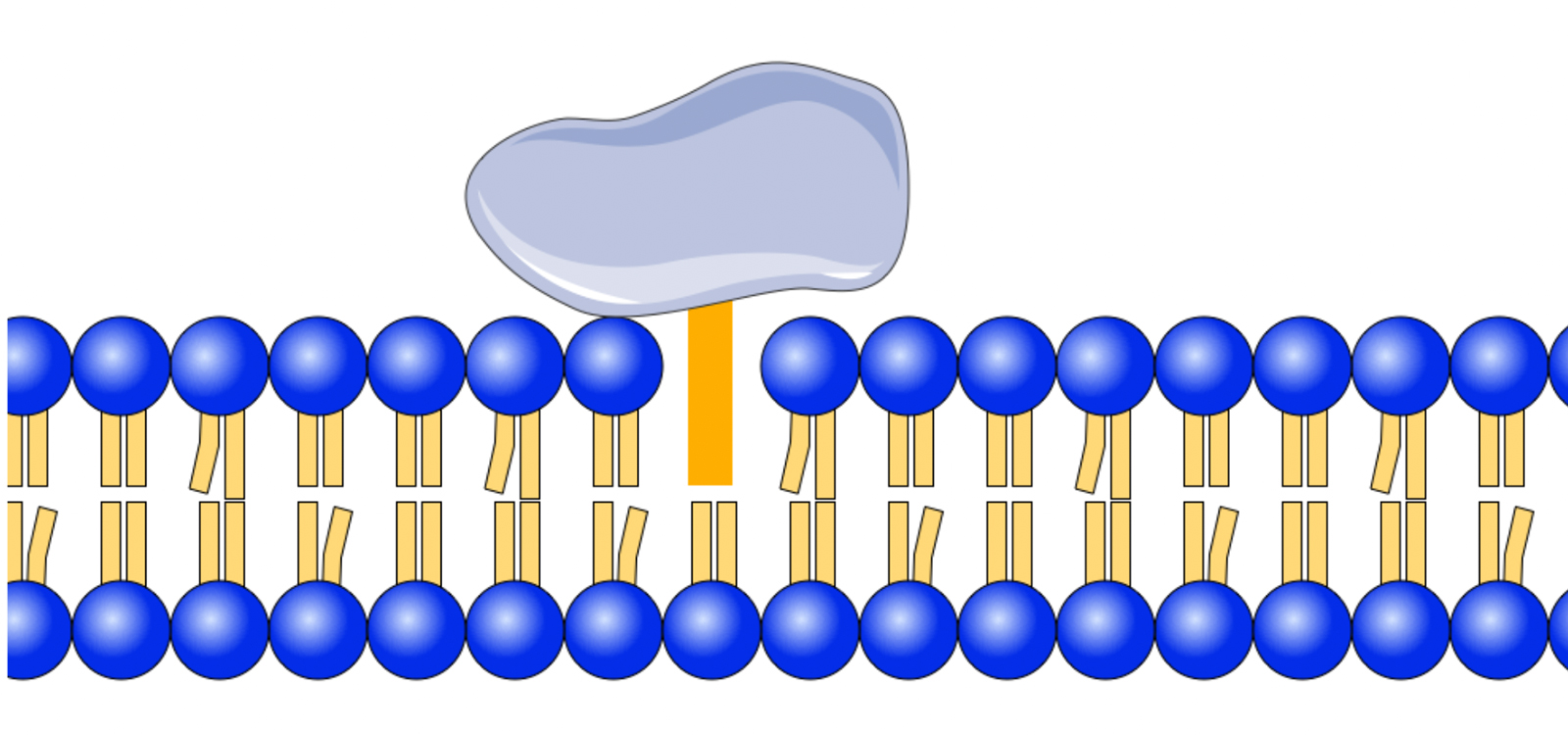
Schema eines Lipid-verankerten peripheren Membranproteins
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Auf dieser Lexikon-Seite sind mehrere Beispiele für Lipid-verankerte Proteine aufgeführt.
GPI-verankerte periphere Proteine
Die "normalen" Lipid-verankerten Proteine (siehe oben) befinden sich alle auf der cytoplasmatischen Seite der Membran. Es gibt aber auch zahlreiche Proteine, die sich auf der extrazellulären Außenseite der eukaryotischen Zellmembran befinden. Viele dieser Proteine sind auf komplexe Weise über Zucker-Einheiten und Fettsäuren in der Membran verankert.
Die Verbindung Glycosylphosphatidylinositol (GPI) spielt eine besondere Rolle bei der Verankerung von Proteinen, man bezeichnet das Lipid, über das das Proteine mit der Lipid-Doppelschicht verankert ist, auch als GPI-Anker.
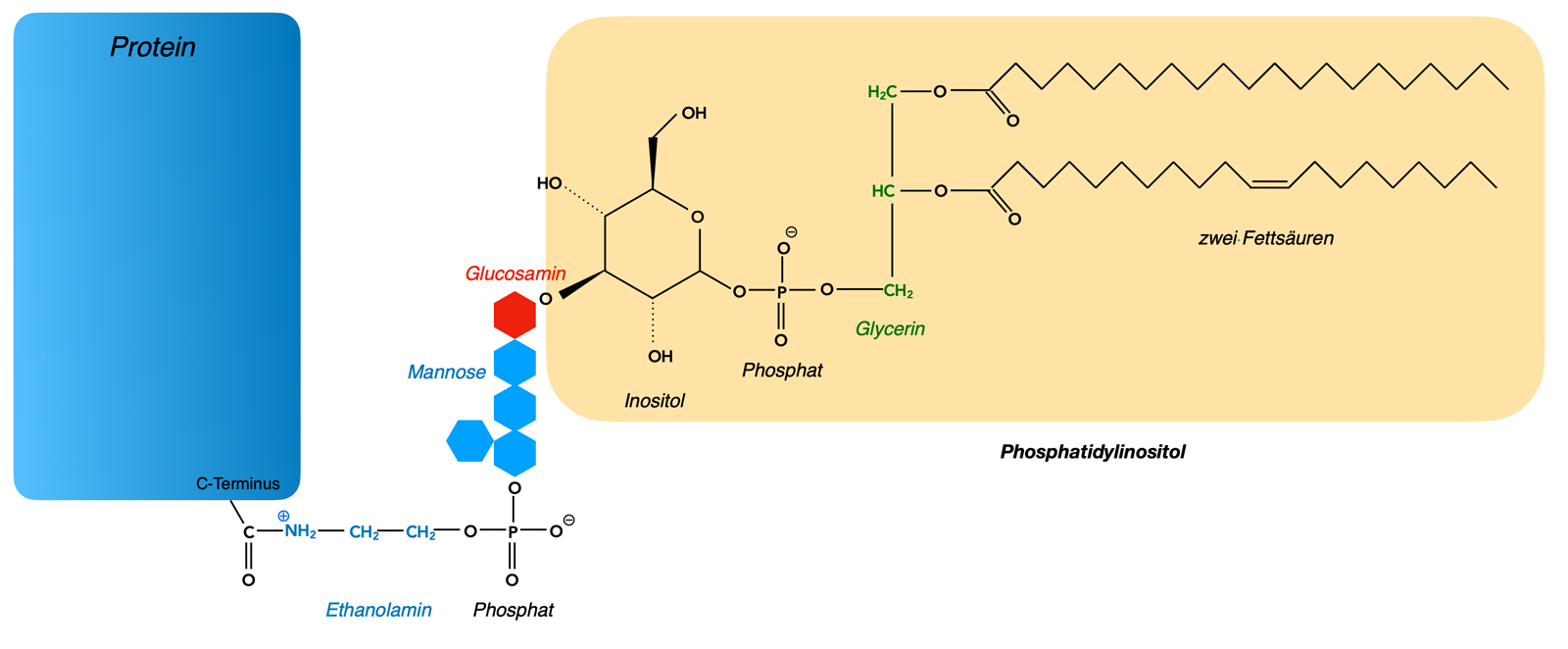
Schema eines GPI-verankerten peripheren Membranproteins
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Auf dieser Lexikon-Seite sind mehrere Beispiele für GPI-verankerte Proteine aufgeführt.
Quellen:
- Stillwell. An Introduction to Biological Membranes. Elsevier Science 2016.
- Nelson, Cox. LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- engl. Wikipedia, Artikel "sodium channel".
- Wikipedia, Artikel "Porine".
- Luckey, Membrane Structural Biology, 2. Auflage, Cambridge University Press 2014.
- engl. Wikipedia, Artikel "Phospholipase A2".
- Deber, Reynolds, "Central nervous system myelin: structure, function, and pathology". Clinical Biochemistry, Vol. 24, Issue 2, April 1991.