Vorbemerkungen
Auf dieser Seite möchte ich kurz die wichtigsten Werkzeuge der Gentechnik vorstellen. Bereits auf der vorhergehenden Seite wurde ein gentechnisches Verfahren beschrieben, bei dem die wichtigsten Werkzeuge eingesetzt werden: Plasmide, Restriktionsendonucleasen etc.Restriktionsenzyme
Bakterien werden ständig von Viren bedroht, die in sie eindringen und ihren Proteinsyntheseapparat zur eigenen Vermehrung missbrauchen.
Die meisten Viren injiziieren dazu ihre eigene DNA in die Bakterienzelle, während die Virenhülle auf der Außenseite des Bakteriums bleibt. Die eingedrungene Viren-DNA programmiert dann den Proteinsyntheseapparat der Bakterienzelle um, so dass
a) die Viren-DNA mehrfach repliziert wird und
b) durch Transkription und Translation der Viren-DNA neue Viren-Hüllproteine sowie ein paar Enzyme entstehen.
Wenn beide Bestandteile der nächsten Virengeneration - DNA und Hüllproteine - synthetisiert sind, finden sich diese Moleküle zu neuen Viren zusammen. Mit Hilfe der synthetisierten Enzyme wird die Bakterienzellwand aufgelöst, und die neuen Viren verlassen die zerstörte Zelle.
Im Laufe der Zeit fand eine immer perfektere Anpassung der Bakterien gegen diese ständige Bedrohung statt. Dabei entstanden eine Reihe von Abwehrmechanismen. Bestimmte Enzyme - die Endonucleasen - waren in der Lage, die eingedrungene Viren-DNA zu zerstören. Dadurch wird eine Restriktion der Virenvermehrung erreicht, was den Namen Restriktionsendonuclease erklärt.
Endonucleasen erreichen dass, indem sie die DNA mitten durch schneiden, während Exonucleasen die DNA von den Enden her abbauen. Wenn ein Bakterium solche Endonucleasen besitzt, besteht natürlich die Gefahr, dass diese Enzyme auch die zelleigene DNA zerschneiden. Dies wird verhindert, indem so genannte Modifikationsenzyme die Schnittstellen auf der Bakterien-DNA durch Methylgruppen maskieren. Die Endonucleasen erkennen die Schnittstellen auf der eigenen DNA nicht mehr und zerschneiden somit nur noch die eingedrungene Fremd-DNA.
Arbeitsweise einer Restriktionsendonuclease
Es gibt eine Klasse von Restriktionsendonucleasen, die sich besonders gut für den Einsatz als Werkzeug der Gentechnik eignen. Diese Enzyme produzieren "sticky ends". Die Abbildung 1 zeigt, was man unter diesem Begriff versteht:
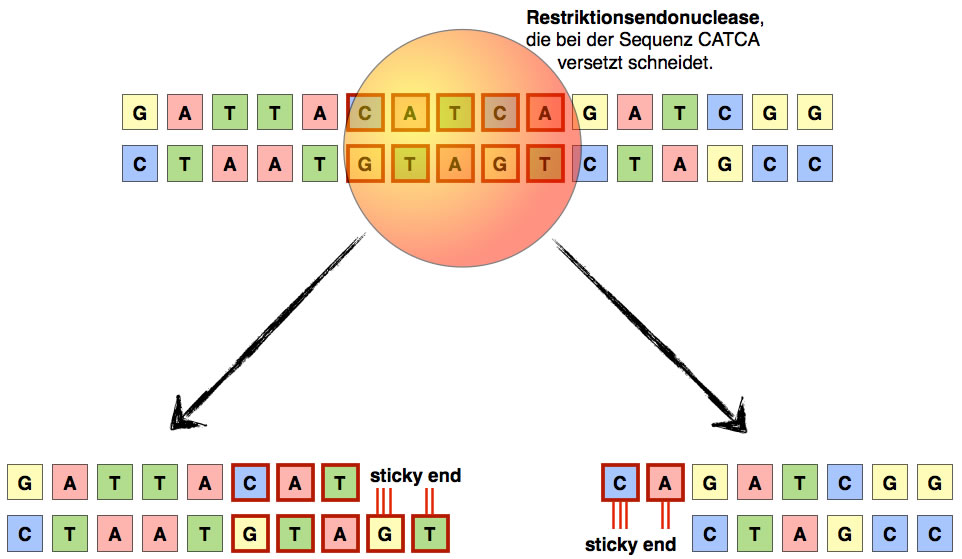
Restriktionsendonucleasen erzeugen sticky ends
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
Das Enzym erkennt eine spezifische Stelle auf der DNA, die in unserem Beispiel durch die Nucleotidsequenz CATCA auf dem oberen Strang gekennzeichnet ist. An dieser Stelle wird die DNA durchtrennt, und zwar so, dass sticky ends entstehen. Darunter vesteht man "klebrige Enden": Die DNA wird nicht glatt durchschnitten, sondern es bleiben einsträngige Bereiche an jedem Ende über. Diese Einstrang-Bereiche können sich nun sehr leicht mit komplementären DNA-Abschnitten paaren und so wieder Doppelstränge bilden.
Was bringen solche sticky ends für die Gentechnik?
Nun, sticky ends verhalten sich ähnlich wie Klebelaschen beim Basteln mit Pappe. Zwei Pappstücke zusammenzukleben, ohne dass sie sich überlappen, dürfte ziemlich schwer sein. Hat eines der Pappstücke aber eine Klebelasche, auf die man dann das andere Pappstück aufsetzen kann, so ist das Zusammenkleben ein Kinderspiel. Eine ähnliche Funktion haben auch die sticky ends beim Zusammenfügen von verschiedenen DNA-Stücken in der Gentechnik, wenn zum Beispiel die Passagier-DNA in das Plasmid eingesetzt werden soll (siehe vorhergehende Seite).
Einbau der Passagier-DNA in das Plasmid
In den nächsten beiden Abbildung wird dargestellt, wie man eine Passagier-DNA in ein Plasmid einsetzt.
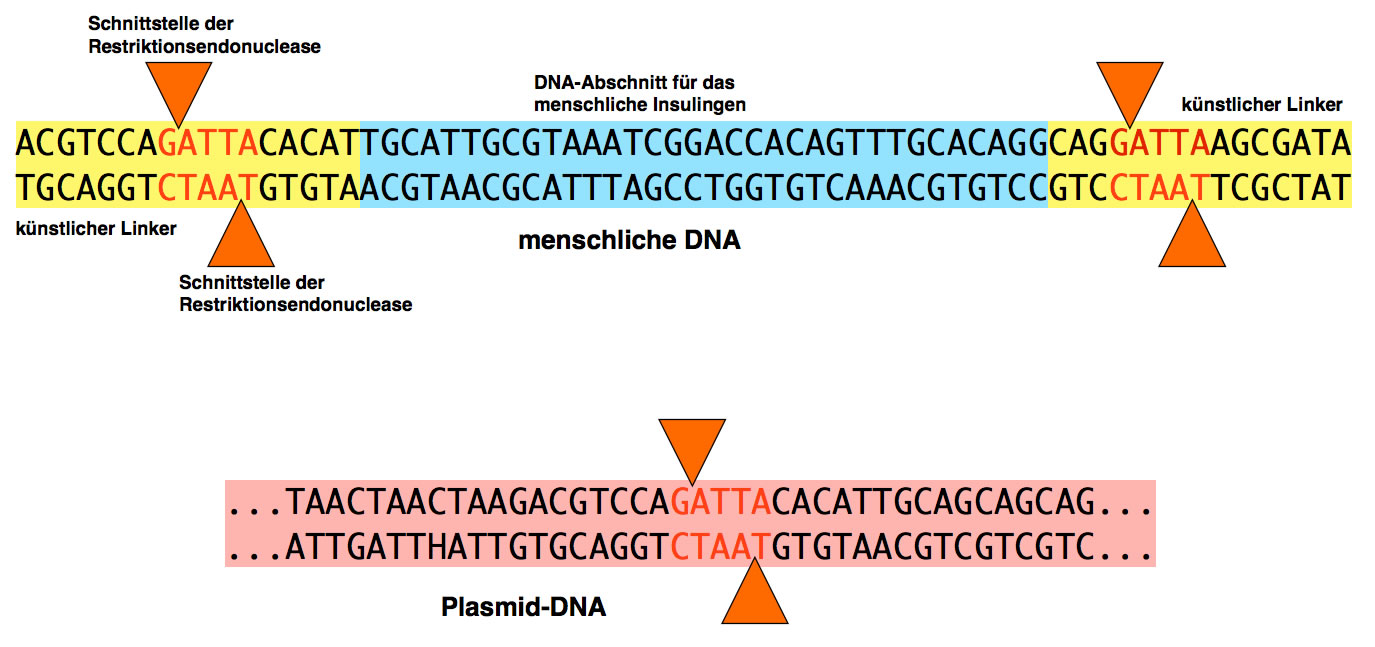
Schnittstellen der verwendeten Restriktionsendonuclease in der Passagier-DNA und in der Plasmid-DNA
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
Die menschliche Passagier-DNA (oben im Bild) hat (künstlich angefügt) eine Schnittstelle für die gewählte Restriktionsendonuclease. Damit die Passagier-DNA in ein Plasmid eingebaut werden kann, muss das Plasmid die gleiche Schnittstelle besitzen.
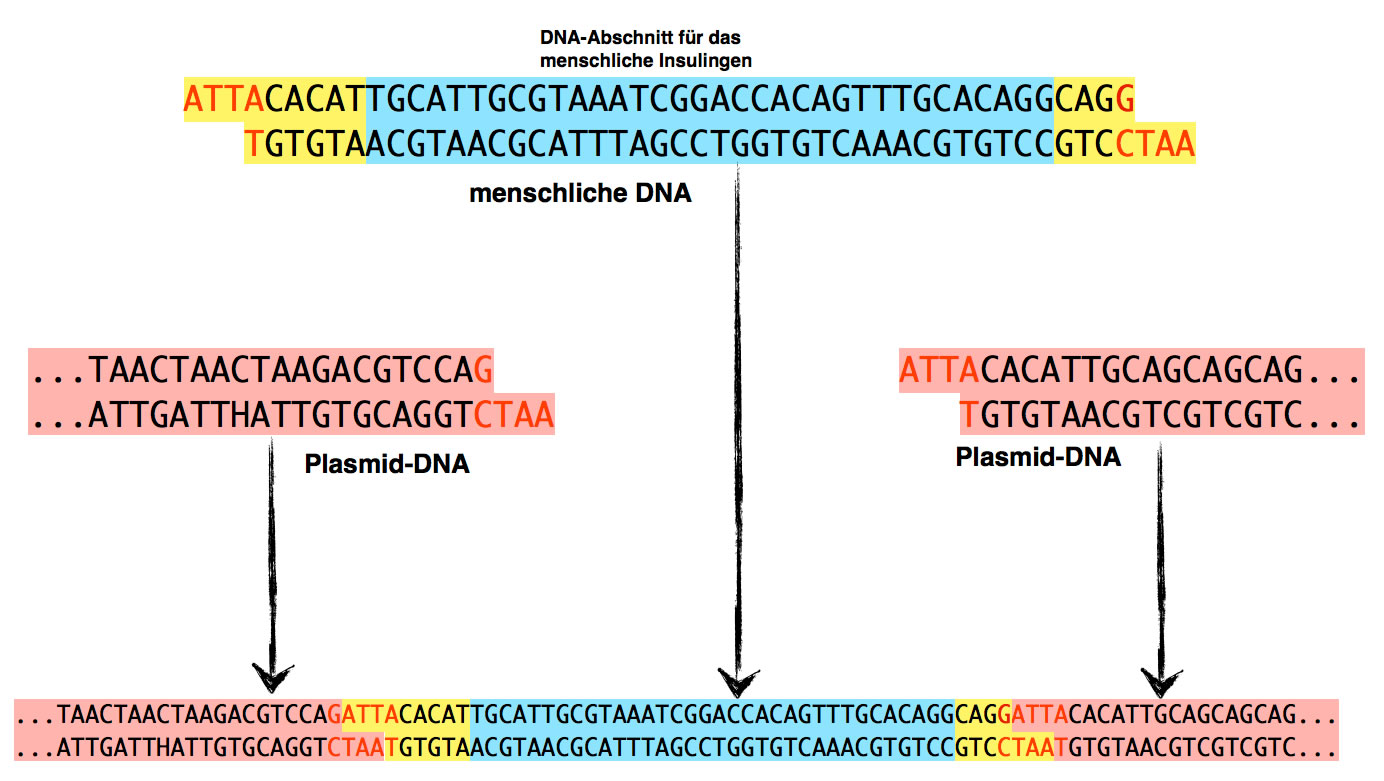
Einbau der Passagier-DNA in das Plasmid
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
Die Restriktionsendonuclease schneidet die Passagier-DNA und das Plasmid so, dass sticky ends entstehen - ideal zum "Zusammenkleben" der beiden DNA-Stränge.
Die Passagier-DNA setzt sich in das geöffnete Plasmid, die beiden DNA-Stränge werden durch die Wasserstoffbrücken zwischen den komplementären Basen der sticky ends zusammengehalten. Das ist noch nicht sehr stabil. Ein weiteres Enzym, eine Ligase, sorgt dann aber dafür, das die Passagier-DNA "richtig" in das Plasmid eingebaut wird, indem auch die Phosphat-Desoxyribose-Grundgerüste kovalent miteinander verbunden werden.
Plasmide
Nicht nur zwischen Bakterien und Bakteriophagen herrscht seit Urzeiten ein Wettrüsten. Die Bakterien sind ebenfalls Krankheitserreger, die in andere Organismen eindringen, zum Beispiel in Pilze. Und diese Organismen haben chemische Abwehrstoffe gegen Bakterien entwickelt, die als Antibiotika bezeichnet werden.
Berühmt ist der Versuch mit den verschimmelten Bakterienkulturen. An den Stellen, wo sich Schimmelpilze gebildet hatten, wuchsen keine Bakterien mehr. So wurde in den 40er Jahren des letzten Jahrhunderts das Penicillin entdeckt. Seitdem hat man viele andere Antibiotika wie Ampicillin, Streptomycin oder Tetracyclin entdeckt. Alle Antibiotika schädigen die Bakterienzelle. Manche stören das Wachstum der Zellwand, andere blockieren Transkription oder Translation oder verhindern die Synthese wichtiger DNA-Bausteine.
Im Laufe der Entwicklung konnte es nicht ausbleiben, dass bei einigen Bakterien bestimmte Enzyme so mutierten, dass sie Antibiotika außer Kraft setzen. Ein Bakterium mit einem solchen Enzym hat einen Überlebensvorteil gegenüber anderen Bakterien. Mit jeder Teilung wird dieser Vorteil an die nächste Generation weitergegeben, bis schließlich, nach wenigen hundert Generationen, nahezu alle Bakterien diese Antibiotika-Resistenz aufweisen.
Mutierte Gene, die eine Antibiotika-Resistenz verursachen, befinden sich auf auf Plasmiden in der Bakterienzelle. Plasmide sind ringförmige DNA-Doppelstränge, auf denen zwei, drei oder vielleicht vier Gene Platz finden. Es können mehrere Plasmide in einer Bakterienzelle existieren, die der Zelle verschiedene neue Eigenschaften verleihen, zum Beispiel Resistenz gegen Tetracyclin.
In Krankenhäusern hat man schon recht bald nach der Entdeckung der Antibiotika festgestellt, dass sich Resistenzen gegen Antibiotika sehr viel schneller ausbreiten, als es eigentlich der Fall sein sollte. Und man hat sogar beobachtet, dass eine Antibiotika-Resistenz von einer Bakterienart auf eine völlig andere Bakterienart überspringen kann. Im Elektronenmikroskop fand man dann die Antwort auf die Frage, wie ein solcher Gentransfer stattfinden kann: Durch Konjugation. Zwei Bakterien können sich zusammenlegen und quasi miteinander paaren: Das eine Bakterium bildet eine aus Proteinen bestehende Röhre aus, die sich in die Zellwand des Partners bohrt. Durch diese hohle Röhre kann dann die Kopie eines Plasmids von der Spenderzelle in die Empfängerzelle gelangen. War vorher nur der Spender gegen Antibiotika resistent, so ist es nachher auch der Empfänger.
Wenn man einen bakteriellen Infekt hat und Antibiotika schlucken muss, so ist es zunächst recht unwahrscheinlich, dass eine Resistenz bei den krankheitserregenden Bakterien auftritt. Gegenüber den körpereigenen "gesunden" Bakterien sind die pathogenen Bakterien in der Minderzahl. Aber die körpereigenen Bakterien werden durch das Antibiotikum ebenfalls stark dezimiert. Nur die wenigen Exemplare, die zufällig schon vorher ein Resistenzgen gegen das verabreichte Antibiotikum hatten (Präadaption), überleben die Antibiotika-Therapie und vermehren sich dann munter. Das wäre ja auch nicht weiter schlimm, da es sich um harmlose bis nützliche Bakterien handelt. Nun kann es aber zur Plasmidübertragung zwischen einem solchen harmlosen Bakterium und einem pathogenen (krankheitserregenden) Bakterium kommen. Und dann sieht es ziemlich schlecht für den Patienten aus, wie man sich leicht ausrechnen kann.
Plasmide sind also etwas durch und durch Natürliches, sie haben sich im Laufe der Zeit entwickelt und den Bakterien einen entscheidenden Vorteil gegen den Abwehrmechanismen der Pilze, Pflanzen und Tiere eingebracht.
Für die Gentechnik eignen sich Plasmide in hervorragender Weise, da sie
- sehr klein sind und künstlich nach Maß hergestellt werden können sowie
- von den meisten Bakterien bereitwillig aufgenommen werden.
Wenn man ein Plasmid für die Gentechnik einsetzen will, so muss es folgenden Anforderungen genügen (Abbildung 4):
- es muss genau eine Schnittstelle für eine bestimmte Restriktionsendonuclease haben. Und zwar für die Restriktionsendonuclease, mit der man das Passagier-Gen aus der Wirts-DNA herausschneiden will.
- es muss einen Replikationsursprung haben, damit es später in den Bakterienzellen repliziert werden kann.
- es muss ein Antibiotika-Resistenz-Gen haben, das durch die Endonuclease nicht zerschnitten wird. Anhand dieses Gens kann man dann später die Bakterien identifizieren, die das Plasmid enthalten.
- es mus ein zweites Antibiotika-Resistenz-Gen haben, welches durch die Endonuclease zerstört wird. Die Passagier-DNA wird also mitten in dieses zweite Gen eingebaut. So kann man später die Bakterien identifizieren, die ein Plasmid mit Passagier-DNA aufgenommen haben.
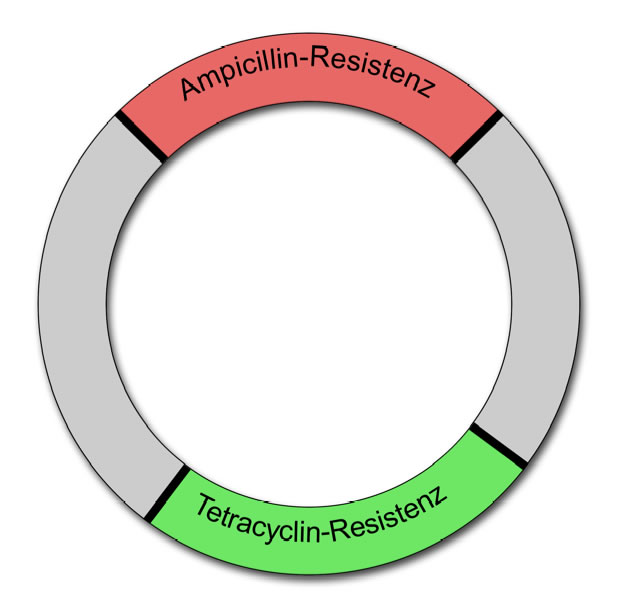
Ein Plasmid, das den oben genannten Anforderungen genügt
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
Gensonden und DNA-Chips
Gensonden sind kurze Stücke einsträngiger DNA (oder RNA), die mit einem Fluoreszenzfarbstoff markiert sind und sich komplementär an einsträngig gemachte DNA oder cDNA anlagern und so den Ort von bestimmten Genen oder anderen gesuchten DNA-Sequenzen markieren.
DNA-Chips sind Mikroarrays, die Tausende verschiedener Gensonden auf kleinstem Raum unterbringen. Mit einem DNA-Chip kann man viele verschiedene Gene in einer Probe gleichzeitig nachweisen.
Auf dieser Lexikonseite werden Gensonden und Genchips näher erläutert.