Initiation - Start der Translation
RNA-Bindung
Als erstes muss sich die mRNA an die kleine Ribosomen-Untereinheit binden. Dazu gibt es auf der mRNA ca. 8 bis 12 Basen vor dem Startcodon eine Ribosomen-Erkennungssequenz, die nach ihren Entdeckern als Shine-Dalgarno-Sequenz bezeichnet wird [2, S. 96]:
5'-GGAGGU-3'
Diese Erkennungssequenz kann sich nun komplementär mit einer entsprechenden Sequenz auf der 16S-rRNA der kleinen 30S-Ribosomen-Untereinheit paaren. Vorher allerdings müssen sich noch drei Initiationsfaktoren an die 30S-Einheit anlagern, die sinnigerweise als IF1, IF2 und IF3 bezeichnet werden. Außerdem bindet als Energieträger ein GTP-Molekül an die 30S-Einheit [2, S. 100].
Erste Aminosäure
Eine fMettRNA setzt sich mit ihrem Anticodon UAC an das Startcodon AUG der mRNA. Es bilden sich Wasserstoffbrücken-Bindungen zwischen den drei Basen des Codons AUG und den drei Basen des Anticodons UAC aus.
Die beiden MettRNAs
Es gibt bei Prokaryoten zwei verschiedene MettRNAs, nämlich die "normale" MettRNA, und die fMettRNA. Die "normale" MettRNA dient zum Einbau der Aminosäure Methionin in die wachsende Peptidkette. Die etwas anders aufgebaute fMettRNA wird nur beim Start der Translation verwendet. Das Methionin am Anfang des Peptids ist kein "normales" Methionin, sondern an die Aminogruppe wurde ein Formylrest "angebaut":
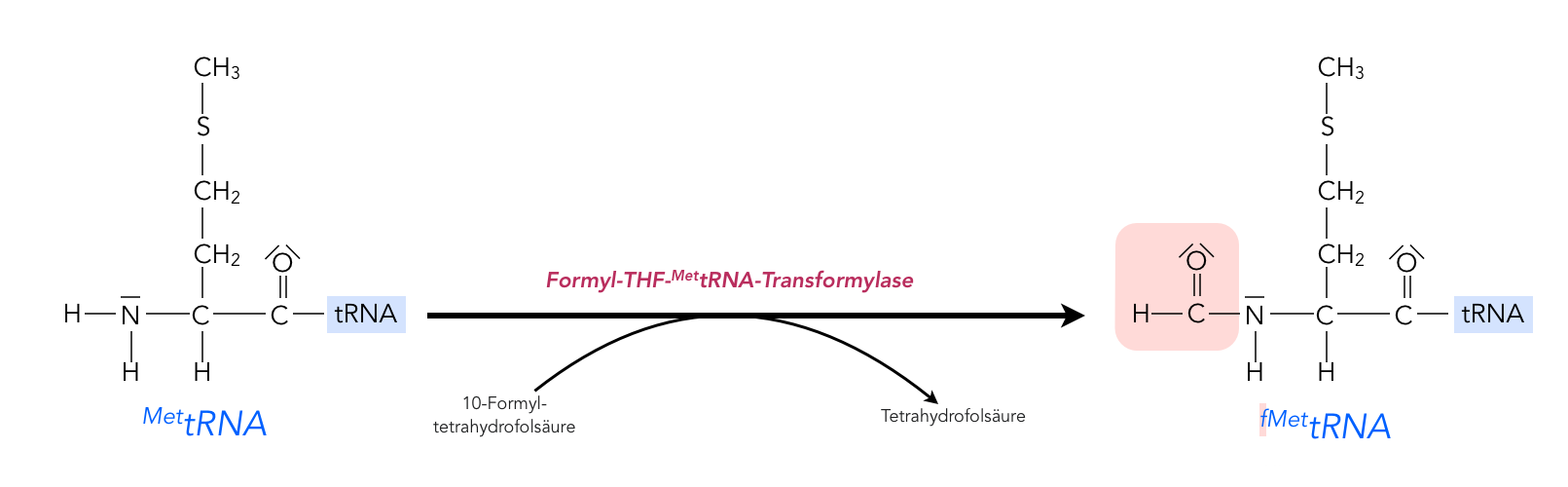
Bildung der fMettRNA
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Diese Reaktion findet erst dann statt, wenn das Methionin bereits mit der tRNA verbunden wurde. Die Formylgruppe (H-C=O, von der Ameisensäure H-COOH bzw. vom Formaldehyd H2C=O abgeleitet) wird von dem Enzym Formyl-THF-MettRNA-Transformylase von einem 10-Formyl-tetrahydrofolsäure-Molekül auf das Methionin-Molekül der MettRNA übertragen [1, S. 79].
Man vermutet, dass durch diese chemische Modifizierung dafür gesorgt wird, dass die nächste Aminosäure nur an die COOH-Gruppe des Methionins angebaut werden kann. Die NH2-Gruppe wird ja durch die Formyl-Gruppe blockiert, hier kann also keine Aminosäure angebaut werden. Nach der Translation, der Fertigstellung des Peptids, wird die Formylgruppe von der ersten Aminosäure wieder entfernt, und zwar durch das Enzym Polypeptid-Deformylase. Oft wird auch das komplette Formyl-Methionin-Molekül entfernt (durch Methionin-Aminopeptidase), denn nicht alle Peptide der Bakterien beginnen mit Methionin [1, S. 78f].
Anlagerung der 50S-Untereinheit
Der Initiationsfaktor IF3 verhinderte bis zu diesem Zeitpunkt die Anlagerung der großen ribosomalen Untereinheit an den Initiationskomplex. Nun fällt dieser IF3-Faktor aber von dem Komplex ab, und die große Untereinheit kann sich anlagern. Die hierfür notwendige Energie wird aus der Spaltung des zuvor gebundenen GTP-Moleküls gewonnen. Die beiden anderen Initiationsfaktoren IF1 und IF2 lösen sich jetzt ebenfalls aus dem Komplex [2, S. 100].
Das Ribosom besteht nun aus den beiden Untereinheiten 30S und 50S und wird als 70S-Ribosom bezeichnet. An der Nahtstelle zwischen den beiden Untereinheiten befinden sich vier wichtige Zentren (bei einem Enzym würde man jetzt von aktiven Zentren sprechen). Drei dieser Zentren dienen zur Aufnahme von tRNA-Molekülen und werden als E-, P- und A-Stellen bezeichnet. Das 'E' steht für "Exitstelle", das 'P' für "Peptidylbindungsstelle" und das 'A' für "Aminoacylbindungsstelle". Die erste tRNA befindet sich nach der Initiation in der P-Stelle. Das vierte Zentrum wird als Peptidyltransferase-Zentrum bezeichnet, dieses Zentrum leistet die "Hauptarbeit" bei der Translation und wird im nächsten Abschnitt (Elongation) näher beschrieben.
Der Initiationskomplex
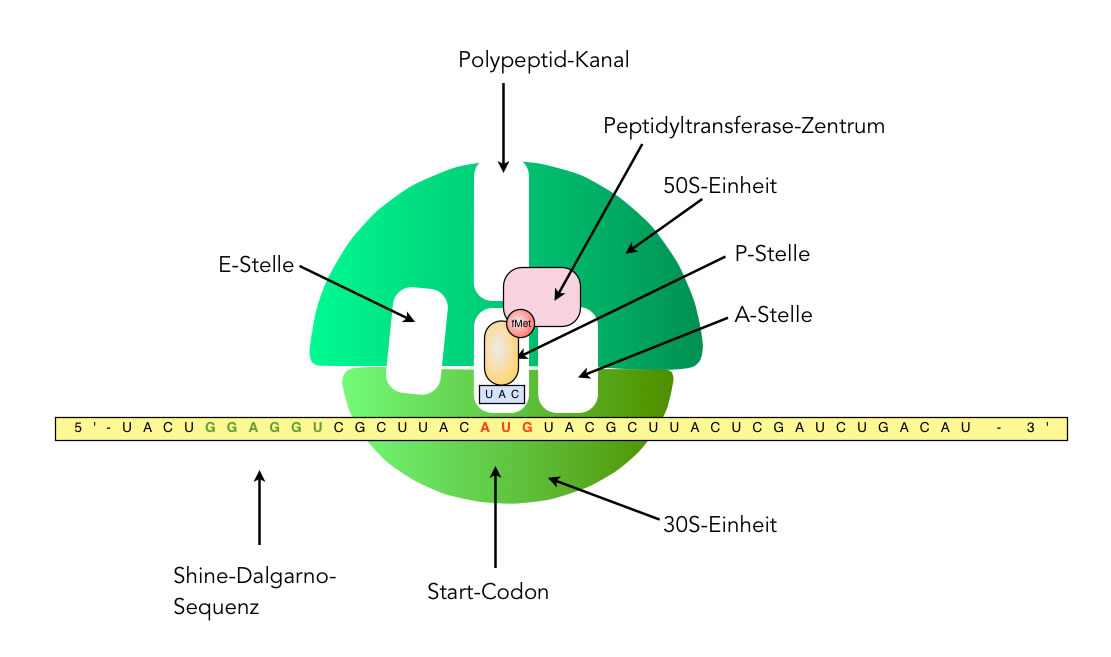
Schematische Darstellung des Initiationskomplexes
Autor: Ulrich Helmich 2022, Lizenz: Public domain
So ungefähr wie in dieser Abbildung könnte man sich den Initiationskomplex der Translation vorstellen [4]. Die P- und die A-Stelle befinden sich in beiden Ribosomen-Untereinheiten, die E-Stelle ebenso. Nur die P- und die A-Stelle gewährleisten der darin befindlichen tRNA den Kontakt zur mRNA. Die tRNA in der E-Stelle hat keinen Kontakt mehr zur mRNA. Das Peptidyltransferase-Zentrum befindet sich in der 50S-Einheit und umfasst die oberen Bereiche der P- und der A-Stelle. Der Polypeptid-Kanal dient zum Ausschleusen des wachsenden Polypeptids.
Elongation
Die Hauptarbeit während der Elongation ist die Übertragung des bisher synthetisierten Peptids auf die neu hinzugekommene Aminosäure durch die Peptidyltransferase. Das bisher hergestellte Peptid ist an die tRNA gebunden, die sich in der P-Stelle des Ribosoms befindet, die neue Aminosäure befindet sich auf der tRNA in der A-Stelle.
Die Peptidyltransferase ist kein Protein, sondern ein RNA-Enzym, ein sogenanntes Ribozym.
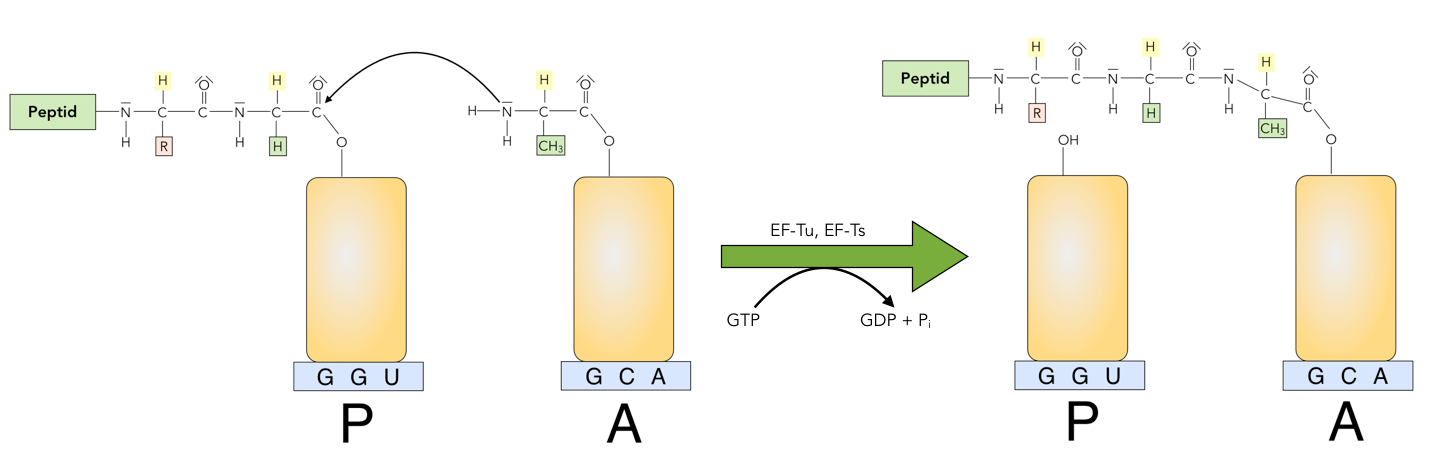
Die Peptidyltransferase-Aktion
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Die Peptidyltransferase-Aktion ist in der obigen Abbildung stark vereinfacht nach [2, S. 98] dargestellt. Chemisch gesehen ist die Reaktion gar nicht so kompliziert, es handelt sich um die Bildung einer normalen Peptidbindung. Das negativ polarisierte N-Atom der neuen Aminosäure greift das positiv polarisierte Carbonyl-C-Atom des bisherigen Peptids nucleophil an.
EF-Tu und EF-Ts sind die Elongationsfaktoren, die an das Ribosom binden müssen, und GTP ist wieder mal der Energielieferant für diese Aktion.
Nach diesem Peptid-Transfer muss das Ribosom drei Nucleotide weiter zum 3'-Ende der mRNA rücken. Dafür ist ein weiterer Elongationsfaktor notwendig, nämlich EF-G. Durch Spaltung von GTP wird dieser Elongationsfaktor aktiviert [2, S. 100].
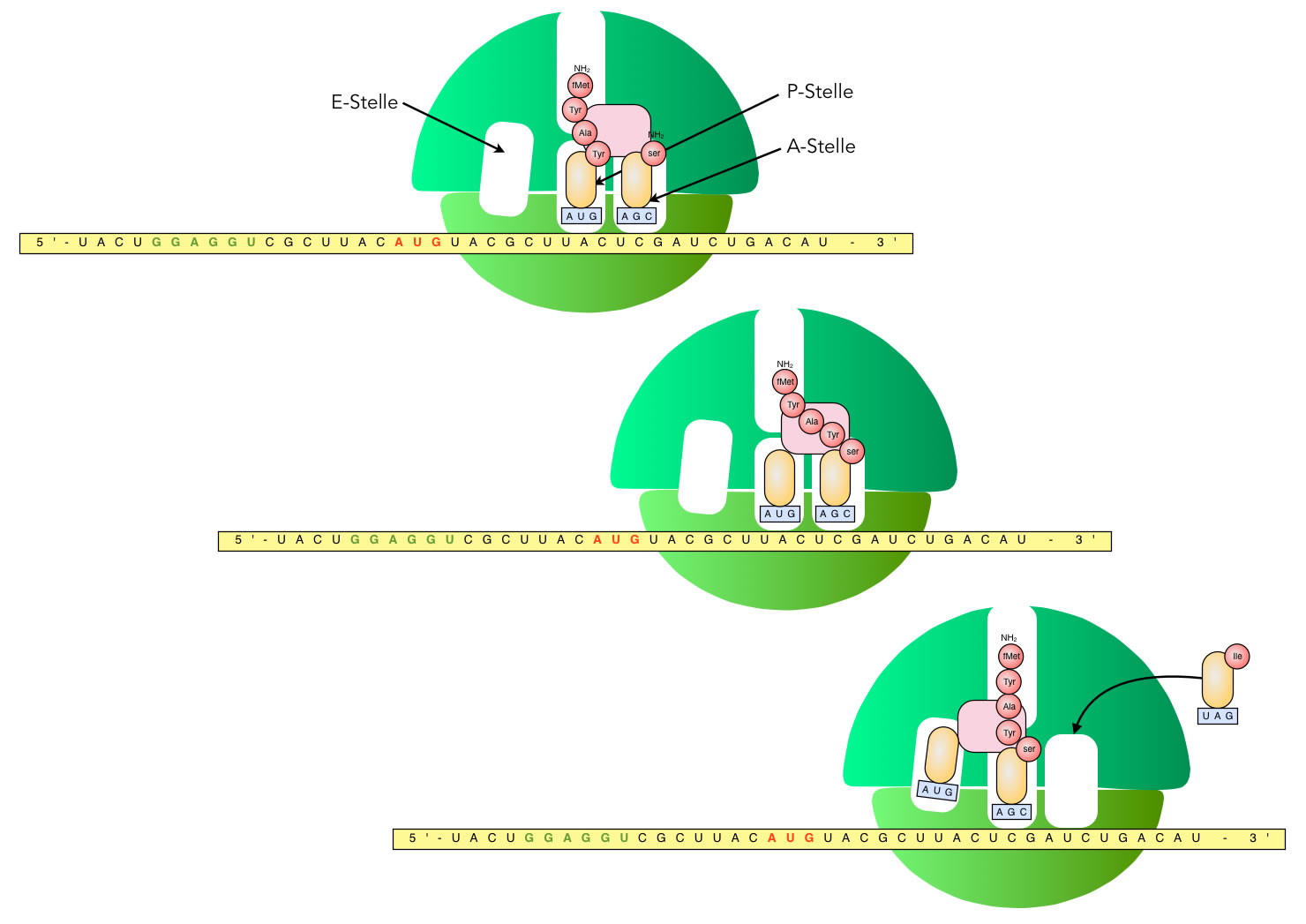
Der Peptid-Transfer und das Weiterrücken des Ribosoms um drei Nucleotide
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Wer es wirklich ganz genau wissen möchte, schaut sich diese Spezial-Seite an.
Das Weiterrücken des Ribosoms um drei Nucleotide führt dazu, dass sich die tRNA der A-Stelle plötzlich in der P-Stelle wiederfindet und die tRNA der P-Stelle in der E-Stelle. Bei dem Platzwechsel von P nach E lösen sich die H-Brücken zwischen Codon und Anticodon auf, die nicht mehr beladene tRNA bereitet sich sozusagen auf den "Exit" vor.
Termination
Die Translation terminiert (wird beendet), wenn ein Stopp-Codon in der A-Stelle des Ribosoms liegt. Für die Codons UGA , UAA und UAG gibt es keine passende tRNA. Der Elongationsmechanismus wird unterbrochen, das Ribosom zerfällt in seine beiden Untereinheiten, und die Translation stoppt. Auch hier spielen wieder andere Faktoren eine wichtige Rolle, die als Terminationsfaktoren bezeichnet werden. RF1 (release factor) erkennt die Stoppcodons UAA und UAG, und RF2 erkennt UAA und UGA. Die Terminationsfaktoren müssen zuvor durch GTP aktiviert werden, dabei spielt der Faktor RF3 eine Rolle, der eine Hilfsfunktion übernimmt [1, S. 89].
RF1 und RF2 haben eine ähnliche Struktur wie eine beladene tRNA und setzen sich in die A-Stelle des Ribosoms. RF3 wird dann nochmals "gebraucht", damit sich RF1 und RF2 vom Ribosom wieder lösen können. Schließlich gibt es noch einen Ribosomenrecyclingfaktor (RRF), der bei der Trennung der Ribosomenuntereinheiten von der mRNA tätig wird[1, S. 89].
Quellen:
- Alfred Nordheim, Rolf Knippers: Molekulare Genetik, 11. Auflage, Thieme-Verlag Stuttgart 2018.
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Nelson, Cox: LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.