Der Transkriptionsvorgang bei Bakterien
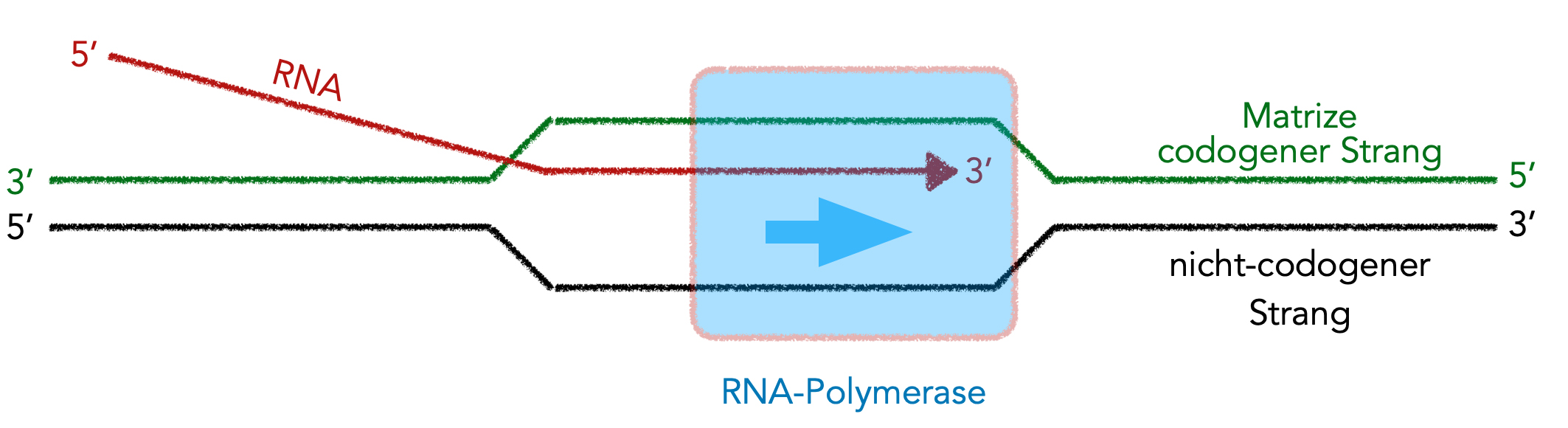
Der Transkriptionsvorgang schematisch dargestellt
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Dieses Bild dient eigentlich nur zur groben Orientierung und soll zeigen, dass die RNA-Polymerase in 3' → 5'-Richtung wandert, vom codogenen Strang aus betrachtet. Genau wie die DNA-Polymerasen hängt auch die RNA-Polymerase die neuen Nucleotide an das 3'-OH-Ende des wachsenden RNA-Strangs. Prokaryoten haben - im Gegensatz zu den Eukaryoten - nur einen Typ von RNA-Polymerasen. Bei Eukaryoten hat man inzwischen fünf verschiedene RNA-Polymerasen gefunden, die unterschiedliche Aufgaben haben.
Sie sollten sich diese Lexikonseite anschauen, bevor Sie weiterlesen. In dem folgenden Text wird nämlich auf die einzelnen Untereinheiten der RNA-Polymerase Bezug genommen. Wenn Sie schon wissen, wie die Polymerase aufgebaut ist, können Sie jedoch auf dieser Seite hier bleiben.
Ähnlich wie die Replikation der DNA besteht die Transkription aus drei Phasen, der Initiation, der Elongation und der Termination.
A. Initiationsphase
Die Initiation der Transkription besteht aus vier Schritten. Zunächst muss die RNA-Polymerase den Anfang des zu transkribierenden Gens finden (1) und sich an das Gen binden (2), dann muss die DNA entwunden auf aufgespalten werden (3), und schließlich muss die erste RNA synthetisiert werden (4). Im Gegensatz zur DNA-Polymerase benötigt die RNA-Polymerase keinen Primer, daher ist der Schritt 4 notwendig.
1. Finden des zu transkribierenden Gens
Die bakterielle RNA-Polymerase bindet "einfach so" an irgendeine Stelle der DNA [1]. Dann gleitet sie an dem DNA-Strang entlang. Die σ-Einheiten der Polymerase versuchen ständig, Bindungen zum DNA-Strang aufzubauen. Sobald die Polymerase einen Promotor erreicht, entstehen festere Bindungen zwischen den σ-Einheiten und bestimmten Stellen des Promotors.
Sie sollten sich auch diese Lexikonseite anschauen, bevor Sie hier weiterlesen. In dem folgenden Text wird nämlich auf die einzelnen Regionen von bakteriellen Promotoren eingegangen. Wenn Sie allerdings schon wissen, was man unter der TATA-Box oder der -35-Region versteht, brauchen Sie diese Seite nicht zu besuchen.
2. Erkennung und Bindung an den Promotor
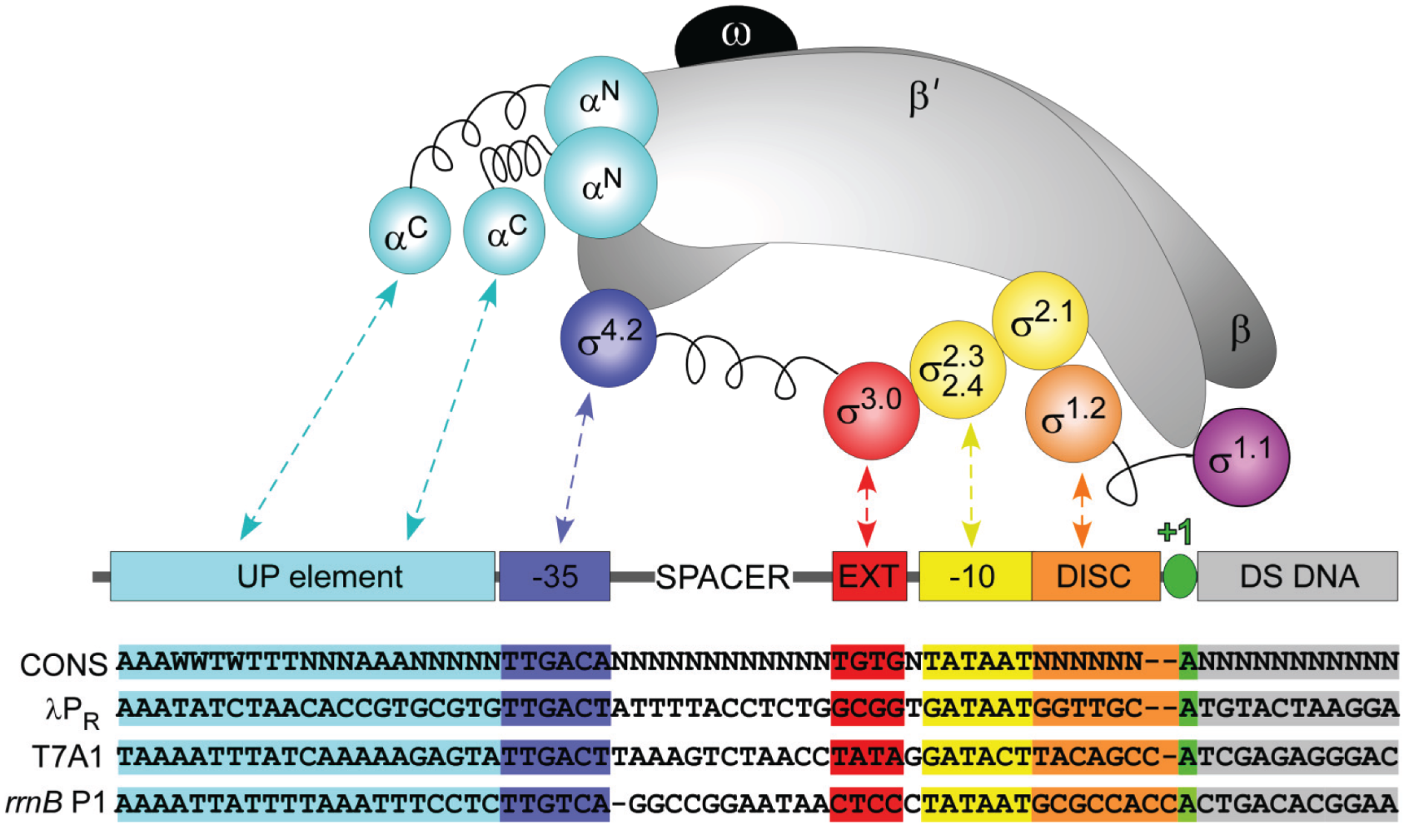
Interaktion der Untereinheiten der RNA-Polymerase mit verschiedenen Promotoren
Quelle: "Initial Events in Bacterial Transcription Initiation" [5], Autoren: Ruff, Records, Artsimovitch 2015, Lizenz: CC BY 4.0
Auf diesem Bild sieht man gut, wie die einzelnen Untereinheiten der RNA-Polymerase mit den verschiedenen Regionen des Promotors interagieren. Die σ-Untereinheiten binden an die TATA-Box an Position -10, eine σ-Untereinheit bindet an die -35-Box, und die C-terminalen Enden der beiden α-Untereinheiten binden leicht versetzt an zwei Stellen des UP-Elements vor der -35-Box.
3. Initiation der Transkription
Die unter 2. beschriebene Situation, also wenn die RNA-Polymerase den Promotor des Gens gefunden hat und sich an den Promotor angehängt hat, wird als geschlossener Promotorkomplex bezeichnet [1, 2]. In diesem geschlossenen Promotorkomplex liegt die Promotor-Region des Gens noch als helixförmiger Doppelstrang vor, die H-Brücken zwischen dem codogenen und dem nicht-codogenen Strang haben sich noch nicht gelöst.
Im nächsten Schritt entsteht der offene Promotorkomplex. Die H-Brücken zwischen den Basen in einer kurzen Region zwischen -12 und +4 um den Startpunkt der Transkription [2] öffnen sich, es entstehen zwei kurze Einzelstränge. "Verursacher" des Aufschmelzens der DNA sind die σ-Untereinheiten der RNA-Polymerase. Eine dieser σ-Untereinheiten, σ2 (in der Abbildung gelb), hat nämlich zwei "Taschen" (vergleichbar mit den aktiven Zentren von Enzymen), in die je eine Base der TATA-Box (in der Abbildung gelb) hineinpasst, nämlich Adenin an Position -11 und Thymin an Position -7 [2]. Man kann sich nun bildlich vorstellen, wie die σ-Einheit an diesen Basen "zieht" und somit die Doppelhelix öffnet. Unterstützt wird dies ja durch die vielen A- und T-Basen in der Promotor-Region, die nur durch je zwei H-Brücken zusammengehalten werden. Eine G- und C-reiche Region wäre viel schwerer aufzulösen.
4. Erste RNA-Synthese
Nun beginnt die Synthese einer kurzen RNA, die vielleicht 10 bis 12 Nucleotide lang ist. Die RNA-Polymerase stellt sozusagen ihren eigenen Primer her.
Die RNA-Polymerase bewegt sich bei dieser Initial-Synthese noch nicht. Manchmal bricht diese Synthese auch wieder ab und beginnt noch mal. Man spricht dann von einer abortiven RNA-Synthese. Wenn aber die kurze RNA eine Länge von 13 bis 15 Nucleotiden erreicht hat, beginnt die Elongationsphase [1].
B. Elongationsphase
In der Elongationsphase wird die kurze Initial-RNA verlängert. Die RNA-Polymerase hängt nach der Vorgabe des codogenen DNA-Strangs (Matrizenstrang) die jeweils komplementären RNA-Nucleotide an das 3'-OH-Ende des bisher synthetisierten RNA-Strangs. Diese Elongation erfolgt in drei Phasen:
1. Bindung des Nucleosidtriphosphats
Das komplementäre Nucleosidtriphosphat, beispielsweise ATP, wird an das aktive Zentrum der Polymerase in der β-Untereinheit gebunden.
2. Anhängen des Nucleotids
Ähnlich wie bei der DNA-Replikation findet nun ein nucleophiler Angriff des O-Atoms der 3'-OH-Gruppe der Ribose auf das Phosphor-Atom der inneren Phosphatgruppe des Nucleosidtriphosphats statt. Es bildet sich eine neue Phosphodiesterbindung zwischen dem neuen Nucleotid und der bisherigen RNA.
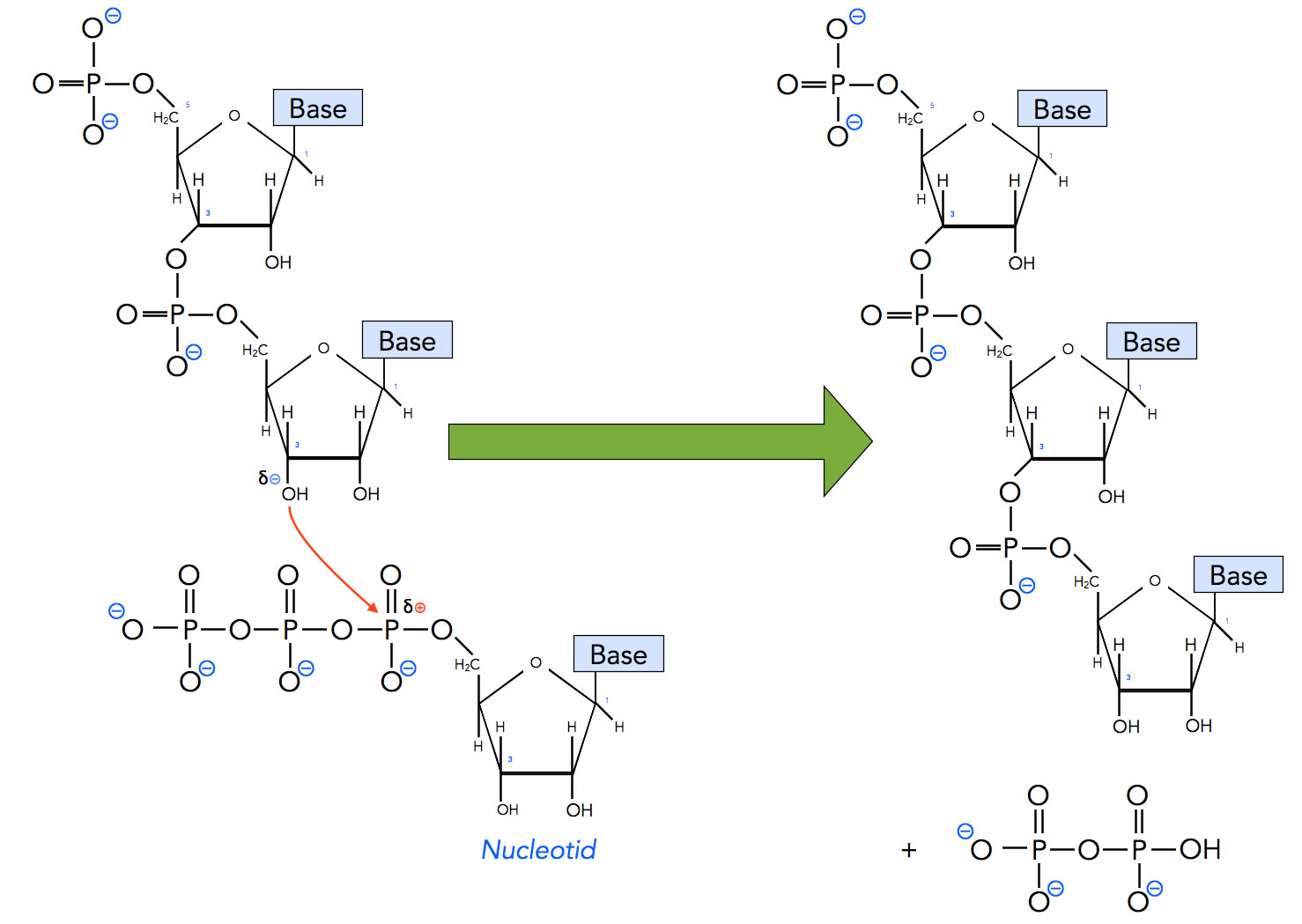
Elongation der RNA
Autor: Ulrich Helmich 2022, Lizenz: Public domain
Dabei wird Pyrophosphat abgespalten, das dann noch in zwei Phosphatgruppen zerlegt wird, was viel Energie liefert.
Noch zwei Anmerkungen:
Ich schreibe hier stets RNA und nicht etwa mRNA, denn bei der Transkription werden ja alle möglichen RNA-Moleküle hergestellt, also auch zum Beispiel die tRNAs oder die ribosomalen RNAs.
Kurze Erläuterung des Begriffs "Phosphodiesterbindung". Die Phosphatgruppen bildet zwei Esterbindungen aus, nämlich einmal mit der 3'-OH-Gruppe der "oberen" Ribose, und einmal mit der 5'-OH-Gruppe der "unteren" Ribose. Daher "Diester".
3. Bewegung der RNA-Polymerase um ein Nucleotid
Wie die Bewegung der RNA-Polymerase auf der RNA genau erfolgt, ist Thema aktueller Forschungen. Aber klar ist, dass bei dieser Bewegung die DNA-Doppelhelix von einer der Polymerase-Untereinheiten Stück für Stück aufgebrochen wird. Die bereits transkribierte DNA weiter stromabwärts wird von einer anderen Polymerase-Untereinheit dann wieder zu einem Doppelstrang zusammengefügt. Topologische Probleme wie bei der DNA-Replikation treten bei der Transkription so gut wie nicht auf, weil ja stets nur ein sehr kurzes Stück der DNA entwunden und aufgespalten werden muss und nicht die gesamte DNA, wie bei der Transkription.
Im Vergleich zur DNA-Polymerase ist die RNA-Polymerase eine "lahme Ente", es werden pro Sekunde vielleicht 60 bis 80 Nucleotide an die wachsende RNA-Kette angehängt [2, S. 80], unter optimalen Bedingungen vielleicht sogar 100 Nucleotide pro Sekunde [1, S. 74]. Bei der Replikation der DNA werden dagegen Geschwindigkeiten von bis zu 1000 Nucleotiden pro Sekunde erreicht - bei Escherichia coli unter optimalen Bedingungen [2, S. 47].
Außerdem kann es immer wieder mal passieren, dass die RNA-Polymerase anhalten muss, weil Nucleotide falsch eingebaut wurden oder Proteine, die an die DNA angelagert sind, den Weg blockieren.
C. Terminationsphase
Bei Bakterien hat man zwei verschiedene Arten der Transkriptions-Termination entdeckt, die rho-unabhängige und die rho-abhängige Termination. Beide Arten der Termination kommen ungefähr gleich häufig vor.
ρ-unabhängige Termination
Während der Elongationsphase gleitet die entstandene RNA durch einen Kanal in einer Untereinheit der RNA-Polymerase. Dieser Kanal ist recht eng, so dass die RNA gerade noch durchpasst. Sollten sich aber innerhalb der RNA irgendwelche Haarnadelschleifen oder andere Unregelmäßigkeiten bilden, passt sie nicht mehr in den Kanal, und die RNA-Polymerase hält an.
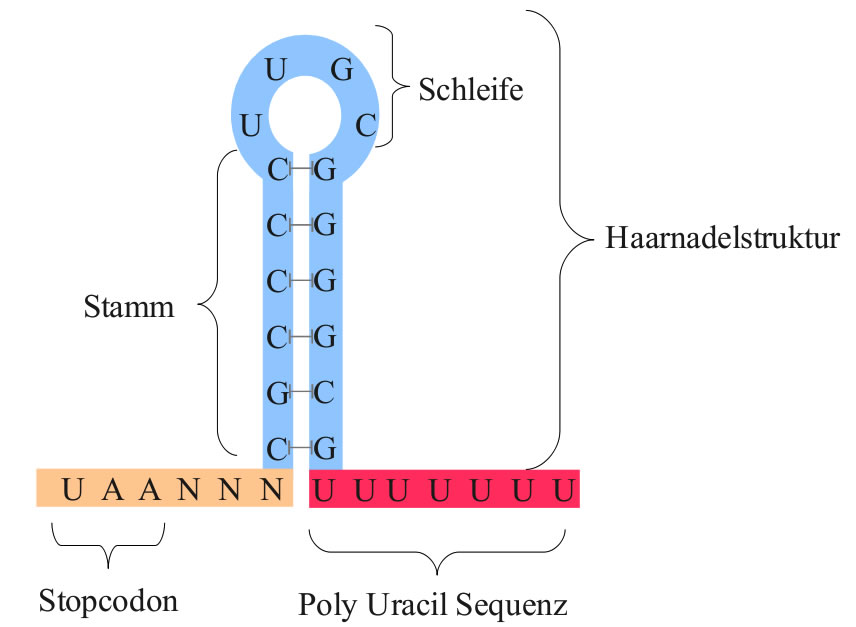
Bildung einer Haarnadelschleife in der RNA beendet die Transkription
Quelle: Wikipedia, Artikel "Terminator (Genetik)", Autor: Rauchbart, Lizenz: public domain.
Genau das passiert bei der rho-unabhängigen Termination. Am Ende der synthetisierten RNA befinden sich viele G- und C-Basen, die so angeordnet sind, dass sich eine Haarnadelschleife bildet, die durch G-C-Basenpaare zusammengehalten wird.
Die RNA-Polymerase hält dann an und fällt von der DNA ab.
ρ-abhängige Termination
Bei der zweiten Terminations-Variante setzt sich ein Terminationsfaktor an einen Bereich der RNA in der Nähe der RNA-Polymerase, der ca. 200 Basen umfasst. Bei dem Terminationsfaktor handelt es sich um das ρ-Protein (Rho-Protein), das aus sechs gleichen Untereinheiten besteht, die einen Ring um die RNA bilden.
Unter Spaltung von ATP bewegt sich das Rho-Hexamer auf das 3'-Ende der RNA zu. Kommt das Protein schließlich bei der RNA-Polymerase an, bewirkt es eine Abspaltung der RNA-Polymerase von der DNA, und die Transkription endet.
Terminator auf der DNA
Bei beiden Formen der Termination spielen Basensequenzen am Ende der hergestellten RNA eine wichtige Rolle. Bei der rho-unabhängigen Termination sind es die vielen G- und C-Nucleotide, die eine Haarnadelschleife bilden, bei der rho-abhängigen Termination ist es eine Erkennungssequenz, an die sich das Rho-Protein anlagert. Die RNA ist aber eine Kopie des codierenden Stranges der DNA, also sind diese Basensequenzen bereits in der DNA enthalten. Diesen Bereich der DNA bezeichnet man allgemein als Terminator. Oft beginnt der Terminator kurz nach dem Code für das Stopp-Codon einer mRNA, so dass das Stopp-Codon noch transkribiert wird.
Quellen:
- Alfred Nordheim, Rolf Knippers: Molekulare Genetik, 11. Auflage, Thieme-Verlag Stuttgart 2018.
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.