Lernziele
Wenn Sie diese Seite durchgearbeitet haben, sollten Sie wissen
- wie die DNA-Replikation bei Prokaryoten verläuft und welche Enzyme welche Aufgaben dabei haben,
- warum die Replikation am Leitstrang relativ unkompliziert verläuft, am Folgestrang jedoch komplexer,
- was man unter Okazaki-Fragmenten versteht und welche Aufgabe die Ligase bei der Replikation hat.
Überblick
Die DNA-Replikation bei Pro- und Eukaryoten kann in drei Abschnitte eingeteilt werden:
- Initiation
- Elongation
- Termination
Auf dieser Seite sollen diese drei Schritte der bakteriellen DNA-Replikation am Beispiel von Escherichia coli kurz erläutert werden, so dass es für das Biologie-Abitur locker reicht.
Wer mehr über die Replikation wissen möchte, vielleicht weil er oder sie Biologie oder Medizin studiert, geht auf die Vertiefungs- bzw. Expertenseiten, auf die im laufenden Text verwiesen wird.
1. Initiation
Die ringförmige Bakterien-DNA (am besten erforscht bei Escherichia coli, dem "Haustier" der Bakteriengenetik) besitzt einen Replikationsursprung, der auch als oriC bezeichnet wird. Bestimmte Proteine, die die Basensequenz von oriC erkennen, lagern sich an die DNA-Doppelhelix und entwinden die DNA ein kurzes Stück, es entstehen zwei kurze DNA-Einzelstränge. Im Elektronenmikroskop kann man zu diesem Zeitpunkt eine kleine Blase in der ringförmigen DNA sehen. Im Zuge der Replikation wird diese Replikationsblase immer größer, bis sich die beiden Replikationsgabeln an den Enden der Replikationsblase auf dem DNA-Ring wieder treffen.
Eine nähere Beschreibung dieses ersten Schrittes der bakteriellen DNA-Replikation finden Sie auf dieser Vertiefungsseite für engagierte LK-Schüler(innen) und Studienanfänger(innen) der Fächer Biologie, Biochemie, Medizin etc.
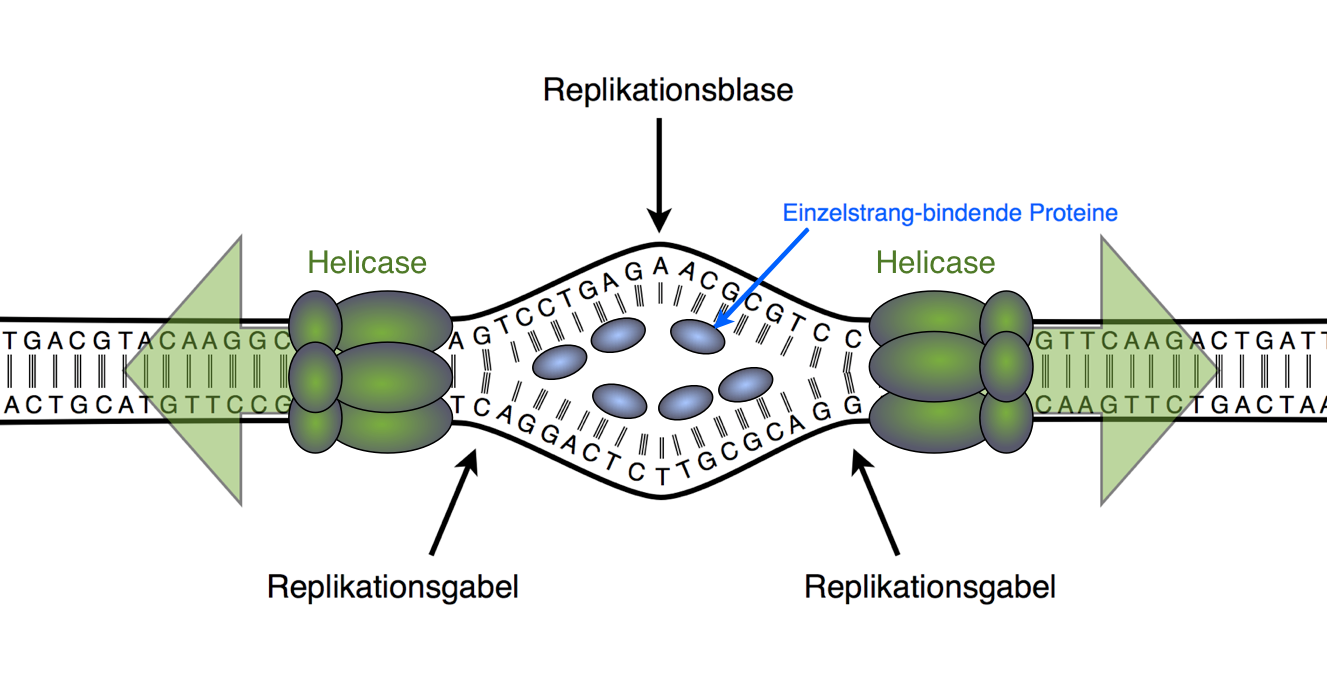
Modellvorstellung einer Replikationsblase
Autor: Ulrich Helmich, Lizenz: Public domain.
Hier sieht man eine solche Replikationsblase im Anfangsstadium. Ein paar der beteiligten Proteine sind eingezeichnet, die anderen Proteine wurden in der Abbildung nicht berücksichtigt (siehe dazu die Expertenseite zur Initiation).
An den Replikationsgabeln entwinden Helicasen den DNA-Doppelstrang. Dadurch entstehen an anderen Stellen der DNA Torsionsspannungen, und Topoisomerasen schneiden das DNA-Rückgrat an bestimmten Stellen durch, so dass sich die DNA wieder "entspannen" kann. Anschließend werden die Schnittstellen wieder verschlossen.
Einzelstrangbindende Proteine lagern sich an die Einzelstränge an und verhindern, dass sich die getrennten komplementären Basen wieder vereinigen. Eine Primase erzeugt ein kurzes RNA-Stück, das zum jeweiligen DNA-Mutterstrang komplementär ist, und die DNA-Polymerase, das Hauptenzym der Elongationsphase, hängt dann neue DNA-Nucleotide an die RNA-Primer.
 |
Die erste DNA-Polymerase wurde von Arthur Kornberg am 16. April 1953 isoliert, er nannte das Enzym DNA-Polymerase I. Kornberg fand auch heraus, dass die Polymerase einen Matrizenstrang benötigt, um zu arbeiten und dass sie die neuen Nucleotide an das 3'-OH-Ende eines DNA-Einzelstrangs anhängt, der dann als Primer bezeichnet wurde. Ohne einen solchen Primer kann die DNA-Polymerase nicht arbeiten. 1959 erhielt Arthur Kornberg den Nobelpreis für Medizin für seine Entdeckungen. Auch sein Sohn Roger erhielt 2006 einen Nobelpreis (Chemie) für seine Arbeiten an der DNA-Polymerase II, und sein anderer Sohn Thomas entdeckte 1970 die DNA Polymerasen II und III. |
2. Elongation
Kontinuierliche Elongation am Leitstrang (leading strand)
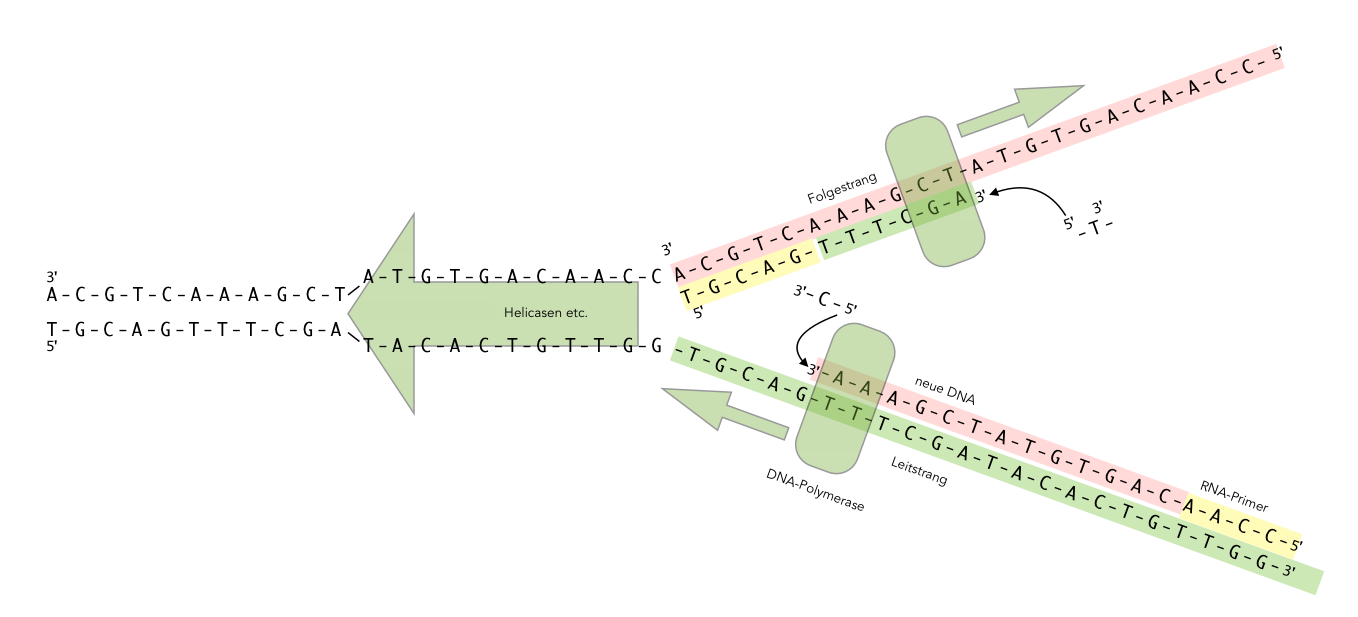
DNA-Synthese am Folgestrang
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Dieses Bild zeigt eine Darstellung der Elongation, wie man sie auch in vielen Schulbüchern der Oberstufe findet.
Rechts unten im Bild sehen wir den sogenannten Leitstrang. Das ist der DNA-Strang, an dem die DNA-Neusynthese ohne Probleme abläuft. Die DNA-Polymerase am Leitstrang folgt der Helicase und den anderen für die Replikation wichtigen Proteinen, die hier nicht weiter erwähnt werden. Auf dem Bild kann man sehen, wie gerade ein Cystein-Baustein mit seinem 5'-Ende an das 3'-Ende der bisher neu synthetisierten DNA angehängt wird.
Ganz rechts am Leitstrang sehen wir einen RNA-Primer. Diese Primer wurde bereits in der Initiationsphase von einer Primase erstellt. Dazu muss man wissen, dass die DNA-Polymerase nicht "einfach so" mit der DNA-Synthese anfangen kann. Es muss bereits ein Einzelstrang vorhanden sein, an dessen 3'-OH-Ende sie dann das nächste Nucleotid anbauen kann. Während der Initiationsphase erzeugt die Primase einen solchen Primer, allerdings besteht dieser nicht aus DNA-Nucleotiden, sondern aus RNA-Nucleotiden.
Diskontinuierliche Elongation am Folgestrang (lagging strand)
Die Elongation am Folgestrang ist etwas komplizierter, weil der Folgestrang sozusagen in die falsche Richtung geht. Auf dem Folgestrang muss sich die Polymerase daher in der Gegenrichtung bewegen, wie man auf dem folgenden Bild gut sehen kann:
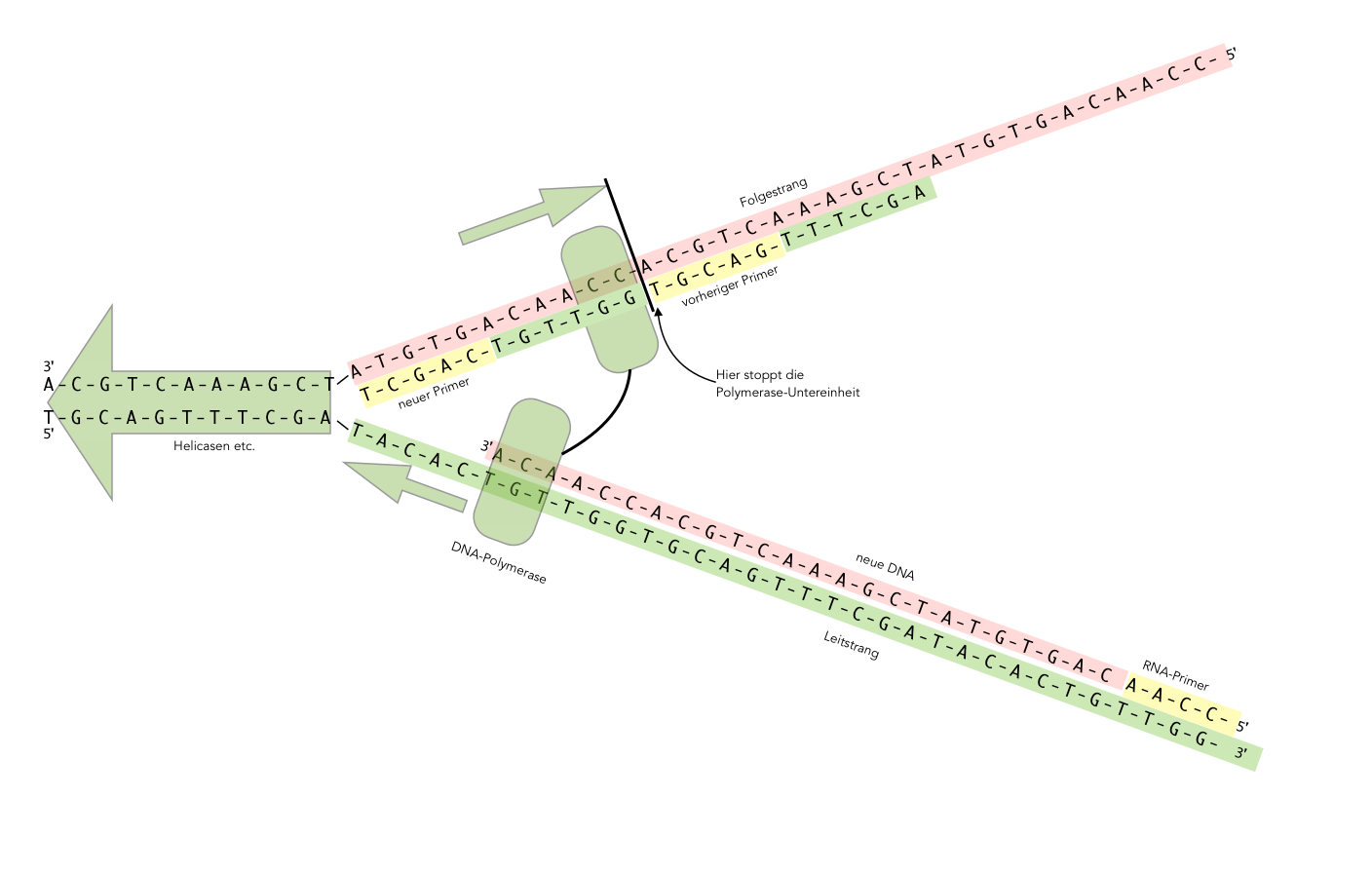
Diskontinuierliche DNA-Synthese am Folgestrang
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Auf der Situation, die in diesem Bild dargestellt ist, hat sich die Helicase weiter nach links bewegt. Die DNA-Polymerase auf dem Leitstrang folgt der Helicase (und den anderen beteiligten Enzymen) in 5'-Richtung.
Auf dem Folgestrang sieht die Situation aber anders aus. Die Polymerase bewegt sich ebenfalls in 5'-Richtung, allerdings in 5'-Richtung auf dem Folgestrang - und das ist genau entgegengesetzt zur Richtung, in der sich die Polymerase auf dem Leitstrang bewegt.
Okazaki-Fragmente
Irgendwann aber erreicht die Polymerase auf dem Folgestrang den vorherigen Primer und stoppt dann mit der DNA-Synthese. Sie löst sich von dem Folgestrang und bewegt sich ein kleines Stück nach links, in Richtung Helicase. Dort hat die Primase inzwischen einen neuen Primer synthetisiert, an den sich die Polymerase nun ansetzen kann, um ein neues kurzes DNA-Fragment zu synthetisieren.
Wenn sie dann auf den vorherigen Primer stößt, löst sich sich wieder von dem Folgestrang und sucht Anschluss an den neuen Primer, der inzwischen wieder gebildet wurde.
Die kurzen DNA-Fragmente, die von der Polymerase auf dem Folgestrang synthetisiert werden, heißen übrigens nach ihrem japanischen Entdecker Okazaki-Fragmente.
Eine Ligase wandert dann über die Okazaki-Fragmente, ersetzt die RNA-Primer durch DNA-Nucleotide und verbindet diese dann zu einem einheitlichen Tochter-DNA-Strang.
Ein kleines Problem gibt es noch
Das war jetzt ungefähr der Kenntnisstand, wie ihn noch viele Oberstufen-Schulbücher vermitteln. Es gibt allerdings noch ein kleines Problem, das nur in wenigen Schulbüchern berücksichtigt wird.
Die eben beschriebene Art der DNA-Synthese mit Okazaki-Fragmenten funktioniert nur, wenn es zwei DNA-Polymerasen gibt, die unabhängig voneinander arbeiten. Die eine folgt dem Leitstrang und arbeitet kontinuierlich, die andere folgt dem Folgestrang und arbeitet diskontinuierlich.
Dummerweise ist es nicht ganz so einfach. Die beiden Polymerase-Untereinheiten sind nämlich relativ fest miteinander verbunden und können sich nicht trennen.
Wie die Natur dieses Problem gelöst hat, soll das folgende Foto verdeutlichen:

Karteikartenmodell einer Replikation
Autor: Ulrich Helmich 2018, Lizenz: Public domain
Ich habe hier mit Papier und Filzstift ein einfaches Modell der DNA-Replikation gebastelt. Die grüne Pappe repräsentiert die doppelte DNA-Polymerase. In der Pappe befinden sich zwei Schlitze, durch die sich je ein DNA-Einzelstrang einfädeln kann. Die aufgemalten Pfeile symbolisieren die Bewegungs- bzw. Transportrichtung.
Der linke Schlitz repräsentiert die Polymerase-Einheit, die sich auf dem Leitstrang bewegt, der linke Papierstreifen steht für diesen Leitstrang.
Der andere Papierstreifen steht für den Folgestrang. Der Folgestrang muss sich jetzt in der gleichen Richtung durch den rechten Schlitz bewegen wie der Leitstrang durch den linken Schlitz. Dazu ist es erforderlich, dass der Folgestrang eine Schleife bildet.
Der rechte Papierstreifen wird in dem Modell also von unten in den Schlitz eingefädelt. Er geht dann ein kurzes Stück hoch, also in die gleiche Richtung wie der Leitstrang. Kurz danach gibt es wieder eine Schleife, und der Folgestrang führt wieder in die entgegengesetzte Richtung zum Leitstrang.
So ähnlich wie in diesem einfachen Modell arbeitet auch die tatsächliche DNA-Replikation. Die DNA-Polymerase besteht aus vielen kleineren Protein-Untereinheiten. Zwei davon sind für die eigentliche Synthese der neuen DNA zuständig, eine Einheit kümmert sich um den Leitstrang, die andere um den Folgestrang, der tatsächlich eine solche Schleife bildet.
Einzelheiten zur Elongation erfahren Sie auf dieser Vertiefungsseite für LK-Schüler(innen) und Studienanfänger(innen).
Hier wird auch kurz auf die Behandlung des Themas in Schulbüchern eingegangen und es wird untersucht, inwieweit das Thema "DNA-Replikation" eine Rolle im Zentralabitur NRW spielt.
3. Termination
Die Prokaroyten-DNA ist ringförmig und hat nur einen Replikationsursprung. Die Replikationsblase erweitert sich im Laufe der DNA-Replikation, und die Replikationsgabeln entfernen sich immer mehr voneinander. Nun ist die DNA aber ringförmig, das heißt, irgendwann treffen die beiden Replikationsgabeln wieder aufeinander und vereinigen sich. Dann ist die Replikation abgeschlossen, und wir haben zwei doppelsträngige DNA-Ringe vor uns, die nicht mehr miteinander verbunden sind. Die Zelle kann sich nun teilen, und jede Tochterzelle bekommt einen dieser DNA-Ringe.
Bei Eukaryoten ist die Sache komplizierter. Eukaryoten haben ja mehrere lange Chromosomen mit je einem sehr langen DNA-Molekül, zumindest nach der Mitose. Ein solches Chromosom hat nicht einen, sondern viele Replikationsursprünge. Es bilden sich also gleichzeitig viele Replikationsblasen, die immer größer werden. Die Replikationsgabeln dieser Blasen wandern aufeinander zu und vereinigen sich dann. Schließlich ist das ganze Chromosom quasi eine einzige Replikationsgabel, und dann trennen sich die beiden DNA-Tochterstränge voneinander. Das Chromosom besteht dann aus zwei Chromatiden, und jedes Chromatid enthält eine komplette DNA-Doppelhelix.
Quellen:
- Allgemeines Schulbuchwissen.
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- "Die Entdeckung der DNA Polymerase" auf www.biomol.com.