Allgemeines
RNA-Viren
In RNA-Viren gibt es RNA-Polymerasen, die die vorhandene Viren-RNA ohne DNA-Vorlage kopieren können.
Pro- und Eukaryoten
Bei prokaryotischen und eukaryotischen Zellen hingegen werden RNA-Polymerasen von der DNA gesteuert. Das bedeutet, dass sie ein Abschnitt der DNA kopieren und basierend darauf eine passende RNA herstellen. Die durch diese Transkription produzierte RNA kann verschiedene Typen umfassen, wie messenger-RNA, ribosomale RNA oder transfer-RNA. Es gibt auch andere Arten von RNA, die in der Genetik der Pro- und Eukaryoten wichtige Funktionen haben.
Prokaryotische RNA-Polymerasen
Während in eukaryotischen Zellen mehrere Typen von RNA-Polymerasen existieren, verfügen Prokaryoten nur über eine einzige RNA-Polymerase. Am Beispiel der bakteriellen RNA-Polymerase soll hier deren Aufbau kurz beschrieben werden.
Die RNA-Polymerase der Prokaryoten besteht aus einem Core-Enzym, das für die eigentliche Transkriptionsarbeit zuständig ist, und mehreren σ-Faktoren, die für die Erkennung der Promotor-Region auf der DNA sorgen.
1. Das Core-Enzym
Das Core-Enzym besteht aus fünf Untereinheiten:
β- und β'-Untereinheit (2 x beta)
Diese beiden beta-Untereinheiten machen die Hauptmasse des Enzyms RNA-Polymerase aus. Die β-Untereinheit besteht aus 1342 Aminosäuren, die β'-Untereinheit sogar aus 1407 Aminosäuren [1]. In der β-Untereinheit ist das aktive Zentrum lokalisiert, in dem die eigentliche RNA-Synthese stattfindet, also das Anhängen neuer RNA-Nucleotide an das 3'-OH-Ende der wachsenden RNA. Die β'-Untereinheit ist ebenfalls an der katalytischen Aktivität der Polymerase beteiligt.
α-Untereinheiten (2 x alpha)
Eine RNA-Polymerase enthält immer zwei α-Untereinheiten, die aus je 329 Aminosäuren bestehen [1]. Das Amino-Ende jeder Untereinheit ist mit den β-Untereinheit verbunden und stabilisiert das Core-Enzym. Das Carboxy-Ende der α-Untereinheiten bindet spezifisch an den Promotor auf der DNA. Außerdem sind die α-Untereinheiten für die Bindung regulatorischer Proteine zuständig.
ω-Untereinheit (1 x omega)
Die ω-Untereinheit ist mit nur 91 Aminosäuren recht klein, trotzdem ist sie wichtig für das Core-Enzym, sie fördert den Zusammenbau und den Zusammenhalt der verschiedenen Untereinheiten.
2. Das Holo-Enzym
Das vollständige Holo-Enzym besteht aus dem oben beschriebenen Core-Enzym sowie mehreren kleineren σ-Faktoren, die an dieses Core-Enzym binden können.
σ-Untereinheiten
Die Ausführungen zu den sigma-Faktoren in der Literatur widersprechen sich teilweise. Einerseits liest man oft, das genau ein sigma-Faktor mit dem Core-Enzym verbunden ist, andererseits ist häufig von bis zu sieben σ-Untereinheiten die Rede, die an das Core-Enzym binden können.
Diese recht kleinen σ-Untereinheiten erkennen bestimmte Startstellen auf dem Promotor der DNA, sie spielen also eine wichtige Rolle bei der Initiation der Transkription. Wenn die Transkription erst mal in Gang gekommen ist, werden die sigma-Faktoren nicht mehr benötigt und lösen sich vom Core-Enzym.
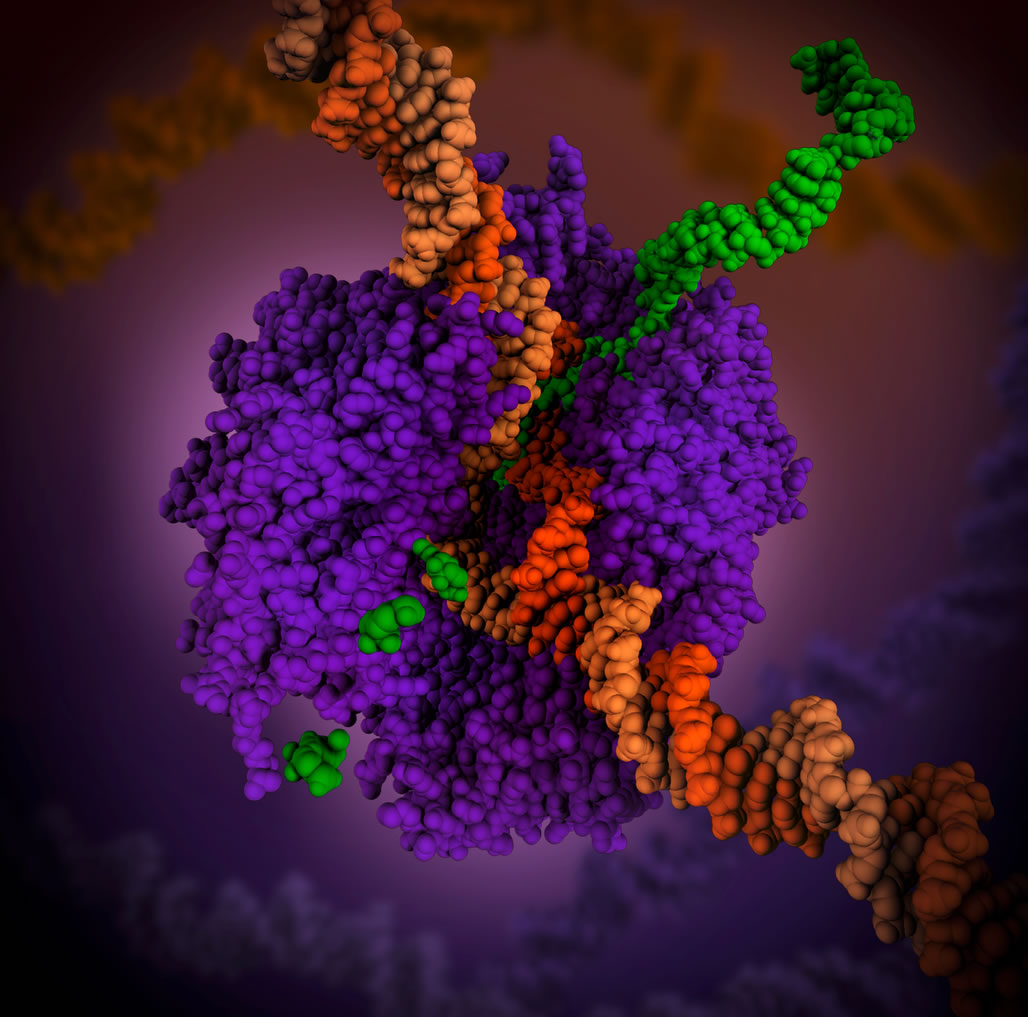
Die RNA-Polymerase in Aktion. Violett: RNA-Polymerase, grün: RNA, dunkelorange: codogener Strang, hellorange: nicht-codogener Strang.
Quelle: engl. Wikipedia, Artikel "RNA polymerase", Autor: Maria Voigt and PDB-101, Lizenz: Creative Commons Attribution 4.0 International license.
Dieses Bild (großes Original-Bild hier) aus der englischsprachigen Wikipedia zeigt sehr schön eine RNA-Polymerase "in Aktion". Die verschiedenen Untereinheiten sind leider nicht farblich hervorgehoben.
Für Experten: die sigma-Faktoren
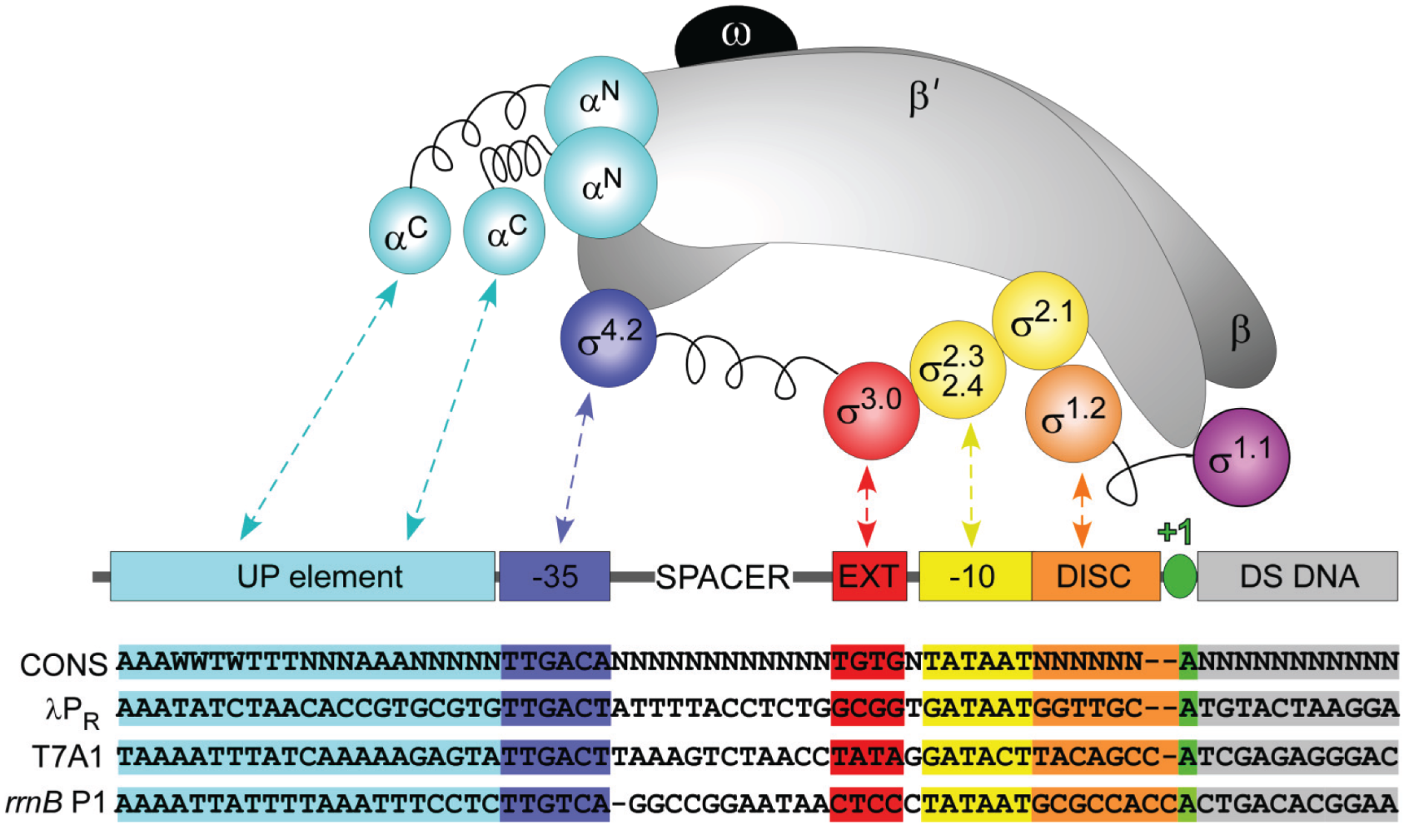
Interaktion der Untereinheiten der RNA-Polymerase mit verschiedenen Promotoren
Quelle: "Initial Events in Bacterial Transcription Initiation" [2], Autoren: Ruff, Record, Artsimovitch 2015, Lizenz: CC BY 4.0
Dieses tolle Bild aus einem Open-Access-Artikel auf der Plattform MDPI zeigt, wie die verschiedenen Untereinheiten der bakteriellen RNA-Polymerase mit bestimmten Regionen von Promotoren wechselwirken. Vor allem die sechs eingezeichneten σ-Faktoren spielen hier eine große Rolle, aber auch die beiden α-Untereinheiten.
Die engl. Wikipedia listet auf der Seite "Sigma factor" die Funktion der verschiedenen sigma-Faktoren recht genau auf. Schauen wir uns diesen Abschnitt des Wikipedia-Artikels näher an:
Specialized sigma factors
- σ70(RpoD) – σA – the "housekeeping" sigma factor or also called as primary sigma factor (Group 1), transcribes most genes in growing cells. Every cell has a "housekeeping" sigma factor that keeps essential genes and pathways operating...
Genes recognized by σ70 all contain similar promoter consensus sequences consisting of two parts. Relative to the DNA base corresponding to the start of the RNA transcript, the consensus promoter sequences are characteristically centered at 10 and 35 nucleotides before the start of transcription (-10 and -35). - σ19 (FecI) – the ferric citrate sigma factor, regulates the fec gene for iron transport and metabolism.
- σ24 (RpoE) – extreme heat stress response and the extracellular proteins sigma factor
- σ28 (RpoF/FliA) – the flagellar synthesis and chemotaxis sigma factor
- σ32 (RpoH) – the heat shock sigma factor, it is turned on when the bacteria are exposed to heat. Due to the higher expression, the factor will bind with a high probability to the polymerase-core-enzyme. Doing so, other heatshock proteins are expressed, which enable the cell to survive higher temperatures. Some of the enzymes that are expressed upon activation of σ32 are chaperones, proteases and DNA-repair enzymes.
- σ28 (RpoS) – the starvation/stationary phase sigma factor
- σ54 (RpoN) – the nitrogen-limitation sigma factor
There are also anti-sigma factors that inhibit the function of sigma factors and anti-anti-sigma factors that restore sigma factor function.
Zusammenfassend kann man also sagen, dass einerseits tatsächlich meistens nur ein σ-Faktor mit dem Core-Enzym verbunden ist, und das ist meistens der "housekeeping" σ-Faktor σ70, der die Konsensus-Sequenzen der meisten Promotoren erkennt.
Unter bestimmten Bedingungen wie Hitzestress, Nahrungsmangel etc. können jedoch auch andere σ-Faktoren mit dem Core-Enzym verbunden sein und die RNA-Polymerase dadurch zu den Promotoren von speziellen Genen leiten, die den ungünstigen Einflüssen entgegenwirken.
Weitere Einzelheiten zu prokaryotischen Promotoren finden Sie auf dieser Lexikon-Seite.
Eukaryotische RNA-Polymerasen
Bei den Eukaryoten gibt es vier verschiedene RNA-Polymerasen, die mit den römischen Ziffern I, II, III und IV gekennzeichnet werden.
RNA-Polymerase I
Diese Polymerase ist für die Synthese ribosomaler RNA verantwortlich, und zwar für die 5,8S-, die 18S- und die 25S-rRNA [3].
RNA-Polymerase II
Das ist quasi die "Schulbuch-RNA-Polymerase". Sie ist nämlich für die Herstellung von mRNA zuständig. Aber auch einige der langen, nicht-codierende RNAs (lncRNAs) werden von dieser Polymerase synthetisiert [3]. Nach [1] werden auch kleine nucleäre RNAs (snRNAs) und mikroRNAs (miRNAs) von dieser Polymerase hergestellt.
RNA-Polymerase III
Diese Polymerase stellt die ribosomale 5S-rRNA und die für die Translation wichtigen tRNAs her [3]. Nach [1] werden auch die 7SL-RNA, die U6-snRNA sowie kleine nucleoläre RNAs (snoRNAs) von der Polymerase III synthetisiert.
RNA-Polymerase IV
Diese Polymerase wurde erst kürzlich entdeckt. Sie ist für die Synthese bestimmter nicht-codierender kurzer RNAs verantwortlich (siRNA, small interfering RNA) [3]. Die Polymerase IV wird im Knippers [1] noch nicht erwähnt, hier ist der Graw [3] eindeutig aktueller. Aber auch der neue Lehninger von 2021 erwähnt die Polymerase IV noch nicht, obwohl schon 2007 ein Artikel "A multistep process gave rise to RNA polymerase IV of land plants" erschienen ist [5]. Immerhin wird diese Polymerase schon in der englischsprachigen Wikipedia erwähnt, wo sogar schon eine Polymerase V aufgelistet ist.
RNA-Polymerase V
Wie bereits erwähnt, listet die engl. Wikipedia bereits eine RNA-Polymerase V auf, die bisher noch in keinem der Hochschul-Fachbücher wie Knippers, Graw oder Lehninger aufgetaucht ist. Auch hier gab es bereits 2008 einen ersten wissenschaftlichen Artikel: "Noncoding transcription by RNA Polymerase Pol IVb/Pol V mediates transcriptional silencing of overlapping and adjacent genes" [6].
Quellen und wichtige Links:
- Alfred Nordheim, Rolf Knippers: Molekulare Genetik, 11. Auflage, Thieme-Verlag Stuttgart 2018.
- Ruff, Record, Artsimovitch: "Initial Events in Bacterial Transcription Initiation" auf www.mdpi.com.
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Nelson, Cox: LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Luo, Hall: "A multistep process gave rise to RNA polymerase IV of land plants", J Mol Evol. 2007 (frei zugänglicher Artikel!).
- Wierzbicki, Haag, Pikaard: "Noncoding transcription by RNA Polymerase Pol IVb/Pol V mediates transcriptional silencing of overlapping and adjacent genes", Cell. 2008 Nov 14; 135(4): 635–648 (frei zugänglicher Artikel!).
- engl. Wikipedia, Artikel "Sigma factor"