Chemische Struktur
Inositoltrisphosphat oder kurz IP3 ist ein Inositol-Molekül, das mit drei Phosphatgruppen verestert ist. Hier die Strukturformel (Wikipedia):
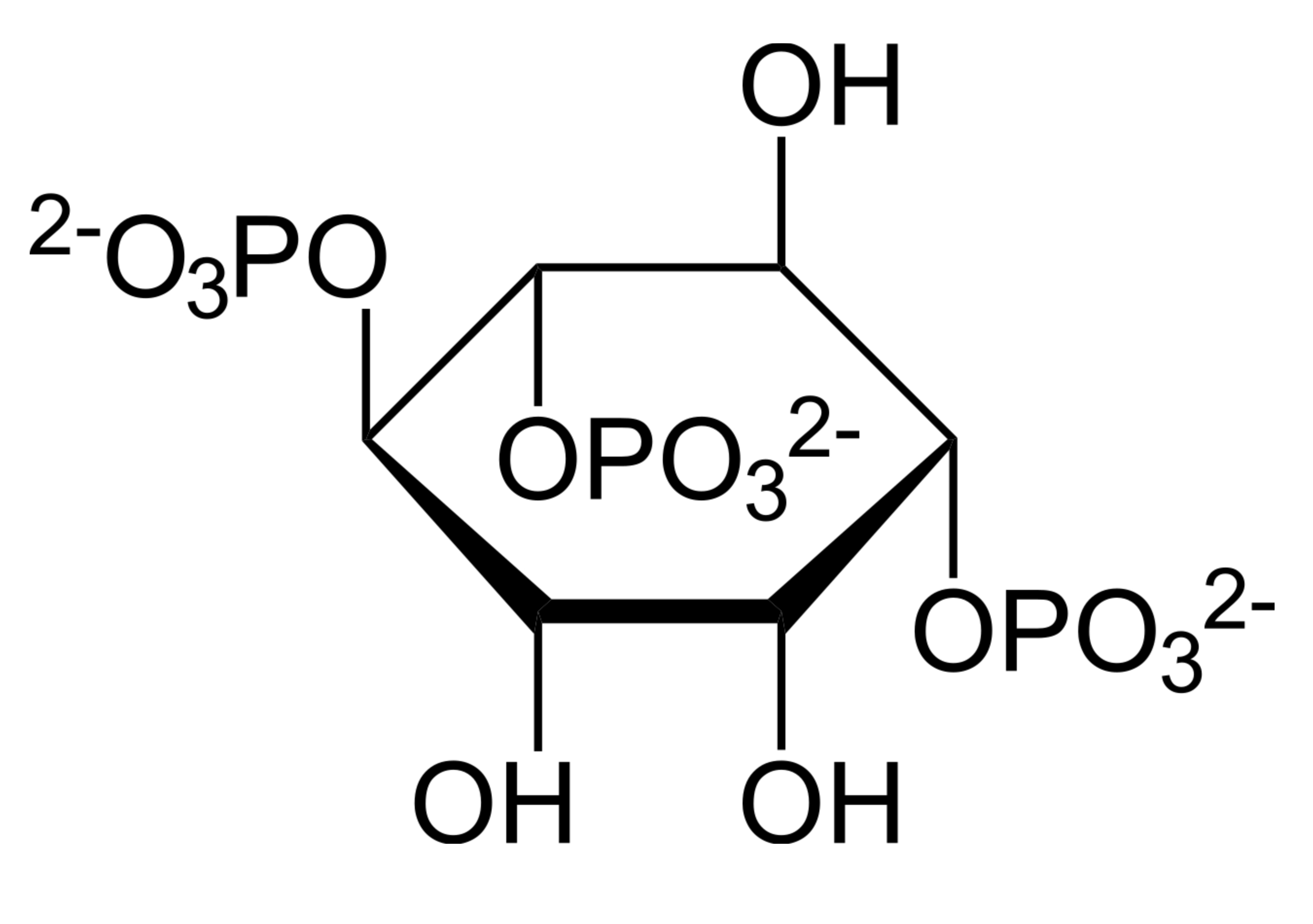
Das IP3-Molekül
Quelle: Wikipedia, Lizenz: Public domain
Biologische Bedeutung
IP3 gehört zu den sogenannten aktiven Lipiden und ist ein second messenger, der frei im Cytoplasma herum diffundiert. Die IP3-Moleküle setzen sich in spezifische Rezeptoren in der Membran des endoplasmatischen Reticulums. Diese Rezeptoren sind mit Ca2+-Kanälen verbunden, die sich daraufhin öffnen. Im ER ist die Ca2+-Konzentration wesentlich höher als im Cytoplasma, daher lösen die IP3-Moleküle einen Ca2+-Ausstrom in das Cytoplasma aus. Ca2+ ist selbst wieder ein second messenger, der zahlreiche andere Proteine aktivieren (oder hemmen) kann.
Es gibt zahlreiche Hormone, die über den second messenger IP3 wirken. Das DocCheck Flexikon [4] listet dabei folgende Hormone auf:
Acetylcholin, Adiuretin, Adrenalin, Angiotensin II, Bradykinin, CCK, Endothelin, Gastrin, Glutamat, Oxytocin, Histamin, Serotonin, TRH und TSH
Bildung von IP3 aus PIP2
Gebildet wird IP3 aus dem Membranlipid Phosphatidyl-Inositol-4,5-biphosphat (PIP2). Durch das Enzym Phospholipase C wird dieses Membranlipid inIP3 und Diacylglycerol (DAG) gespalten, wie man auf dem nächsten Bild sieht:
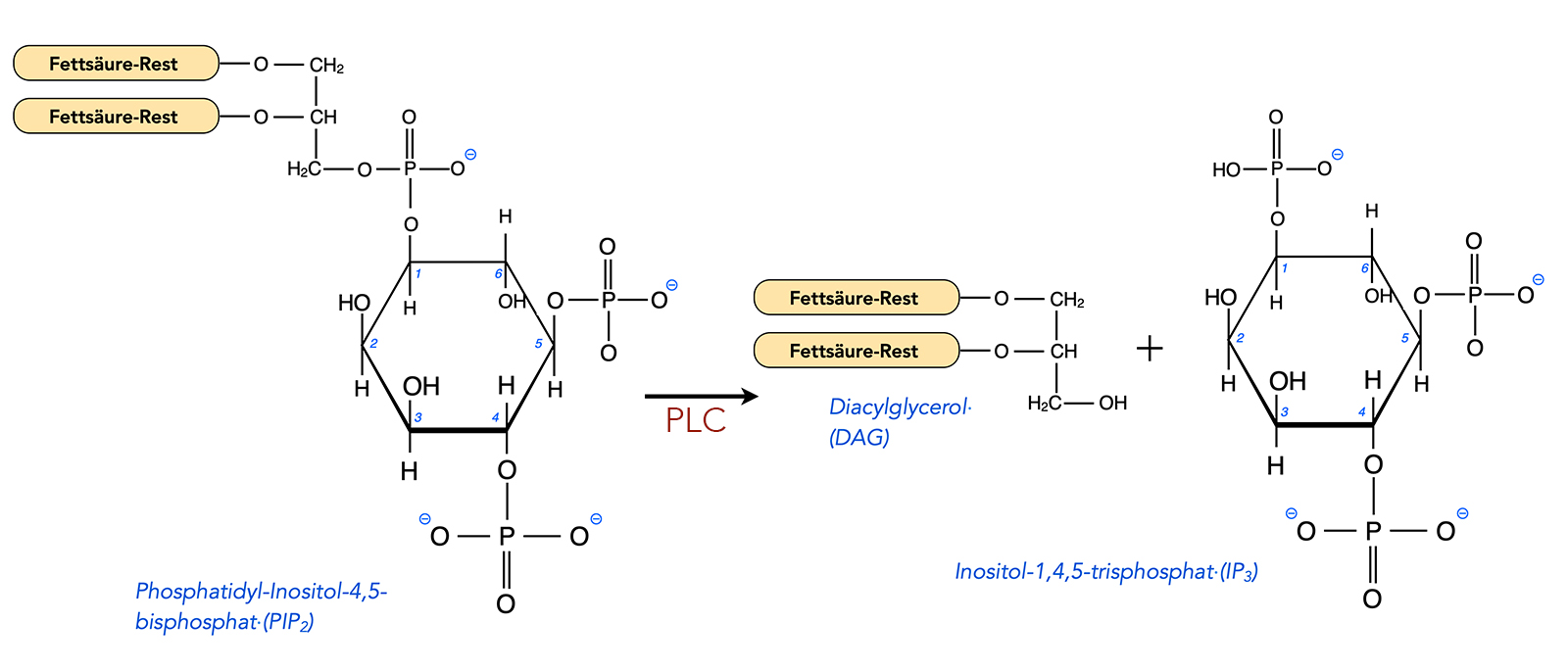
Phospholipase C spaltet PIP2 in IP3 und DAG
Autor: Ulrich Helmich 2022, Lizenz: Public domain.
Auch DAG ist ein wichtiger second messenger.
Die Phospholipase C wiederum wird von einem G-Protein aktiviert, und diese wiederum von einem membrangebundenen Rezeptorprotein.
Deaktivierung von IP3
Ein second messenger muss auch wieder deaktiviert werden können, damit die Zelle auf neue Signal-Moleküle reagieren kann, die einen Anstieg der second-messenger-Konzentration zur Folge haben.
IP3 wird durch das Enzym IP3-Phosphatase zu Inositolbissphosphat IP2 deaktiviert [5].
Für Experten
Das folgende Bild aus der Wikipedia zeigt sehr schön
a) die Aktivierung der Phospholipase C durch einen G-Protein gebundenen Rezeptor
b) die Bildung von IP3 als Folge dieser Aktivierung
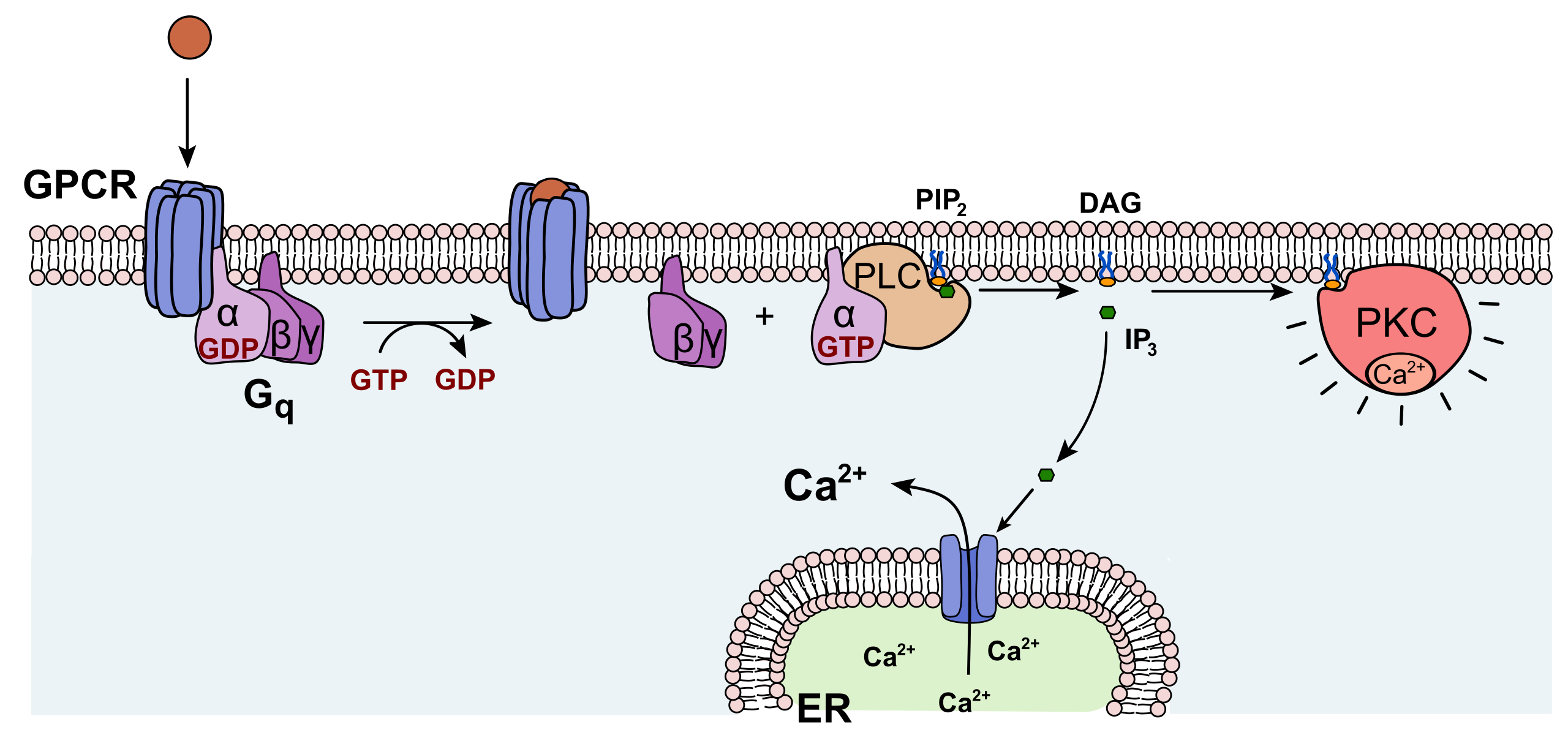
Aktivierung der Phospholipase und deren Auswirkungen
Yikrazuul, CC BY-SA 3.0, via Wikimedia Commons
- Der G-Protein-gebundene Rezeptor wird durch ein Signal-Molekül aktiviert.
- Das GDP-Molekül des G-Proteins wird durch ein GTP-Molekül ersetzt.
- Das G-Protein trennt sich jetzt in seine drei Untereinheiten: α-Einheit/GTP und β/γ-Einheit.
- Der α-Einheit/GTP-Komplex aktiviert nun die Phospholipase C (PLC) auf der Innenseite der Zellmembran.
- Die aktivierte Phospholipase C hydrolysiert nun das Membranlipid Phosphatidyl-Inositol-4,5-biphosphat (PIP2) zu Inositol-1,4,5-triphosphat (IP3) und Diacylglycerol (DAG) .
- IP3 iffundiert nun durch das Zellplasma und setzt sich in spezielle Ca2+-Kanäle im endoplasmatischen Reticulum. Diese öffnen sich und entlassen Ca2+-Ionen in das Zellplasma. Durch den rasch ansteigenden Ca2+-Spiegel werden nun weitere Proteine aktiviert und entsprechende Reaktionen ausgelöst.
Bei dem Wikipedia-User Yikrazuul möchte ich mich ganz herzlich für diese hervorragende Abbildung bedanken, die sicherlich sehr viel Arbeit gemacht hat.
Quellen:
- Alberts et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- Urry, Cain, Wassermann, Minorsky, Reece, Campbell Biologie, Hallbergmoos 2019, 11.Auflage
- Nelson, Cox. LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- DocCheck Flexikon, Artikel "IP3-Signaltransduktion"
- Wikipedia, Artikel "Inositoltrisphosphat"