Als α-Helix oder alpha-Helix bezeichnet man in der Biochemie eine geordnete Sekundärstruktur (ein Faltungsmuster) innerhalb eines Peptids oder Proteins.
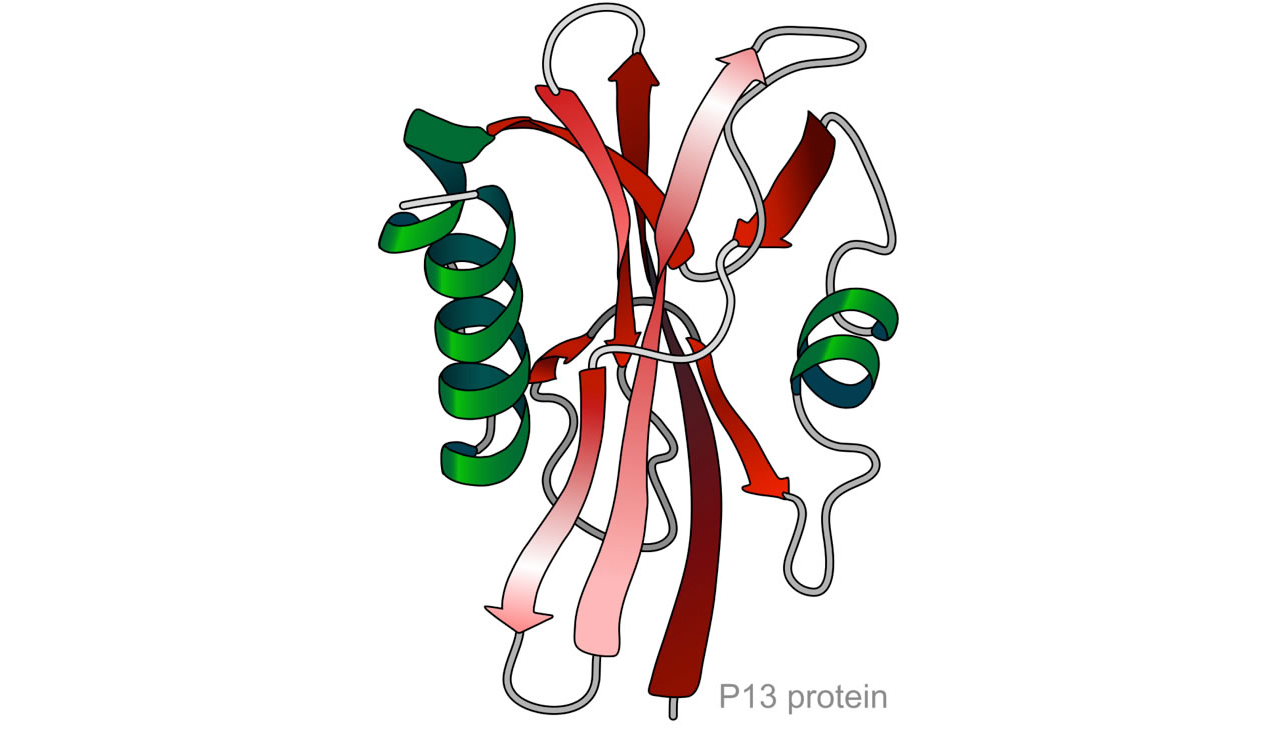
Die Tertiärstruktur eines kleinen Proteins
Quelle: Wikipedia, Artikel "Protein structure", abgerufen im Mai 2013, Autor: nicht mehr bekannt, Lizenz: public domain.
Hier sehen wir die Tertiärstruktur eines kleinen Proteins mit mehreren geordneten Faltungsmustern, den Sekundärstrukturen. Das Protein enthält neben einem großen β-Faltblatt-Bereich (rot gezeichnete Bänderstruktur) zwei α-Helices (grün gezeichnete Spiralstrukturen).
Schon in den 30er Jahren des 20. Jahrhunderts stellte William Astbury (1889-1961) bei seinen Röntgenstruktur-Untersuchungen an kristallisierten Proteinen fest, dass viele Proteine regelmäßige Bereiche enthalten. Der Begriff der Wasserstoffbrücken-Bindung war zu der Zeit schon bekannt, und Astbury nahm an, dass solche H-Brücken an der Bildung dieser regelmäßigen Bereiche beteiligt sind [2].
Linus Pauling hatte 1948 den entscheidenden Durchbruch. Als er Gastprofessor an der Oxford Universität war, wurde er krank und musste einige Tage in seinem Apartment verbringen. Er hatte dort nichts Vernünftiges zum Lesen, also setzte er sich aus purer Langeweile an den Schreibtisch und begann, mit Bleistift und Papier Strukturen für Peptide zu zeichnen, die eine maximale Stabilität besaßen. Dabei kristallisierte sich eine Helix-Struktur heraus. Zusammen mit Robert B. Corey und Herman Branson entwickelte Pauling dann nach weiteren Überprüfungen 1951 das Modell der α-Helix [1].
Bau einer alpha-Helix
In diesem Kasten wird gezeigt, wir man mit einfachen Mitteln selbst eine solche alpha-Helix bauen kann. Gut geeignet (und auch mehrfach getestet) ist dieses Verfahren für den Schulunterricht in der Sekundarstufe II.
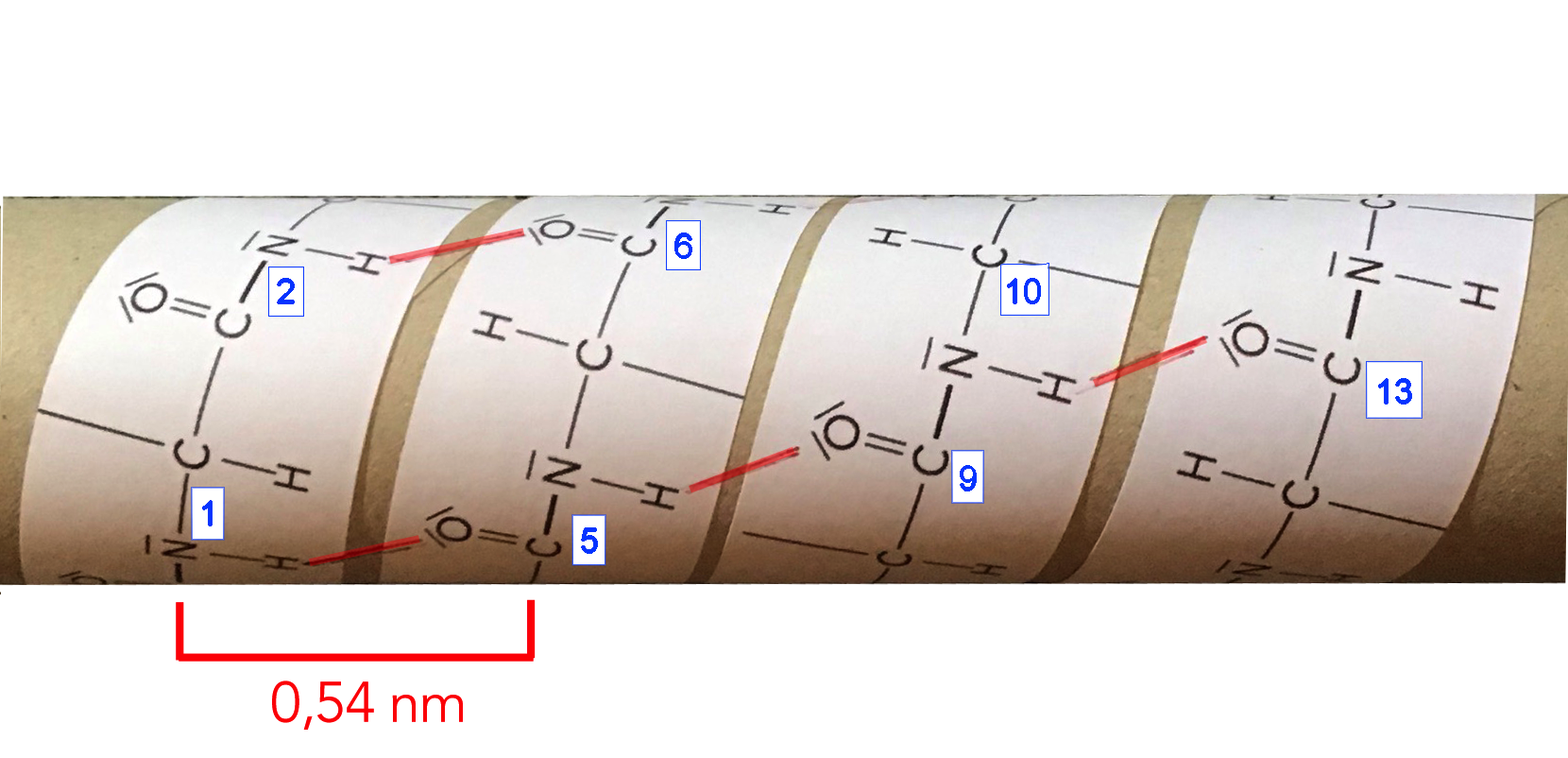
Eine selbst gebastelte alpha-Helix
Autor: Ulrich Helmich, Lizenz: Public domain
Zunächst erstellt man am PC ein langes Peptid mit 15 bis 20 Aminosäuren. Die zwei oder drei Seiten werden dann ausgedruckt, und dann werden die Peptidfragmente ausgeschnitten und zusammengeklebt, so dass sich ein langes Peptidband ergibt.
Dieses Peptidband wickelt man dann um eine geeignete Röhre. Bewährt hat sich beispielsweise die innere Pappe einer Haushaltspapierrolle. Das Peptidband muss so um die Röhre gewickelt werden, dass sich eine spiralige Struktur ergibt, so wie auf dem Photo oben. Außerdem muss darauf geachtet werden, dass eine Windung der Helix 3,5 bis 4 Aminosäuren umfasst (der Idealwerte wäre 3,6).
Dann klebt man das Peptidband auf die Pappröhre auf und verbindet das Stickstoff-H-Atom der Aminosäure 1 mit dem Carbonyl-O-Atom der Aminosäure 5 mit einem roten Strich, der eine H-Brücke symbolisieren soll. Entsprechend verfährt man mit den Aminosäuren 2 und 6, 3 und 7 und so weiter.
Die Seitenketten der beteiligten Aminosäuren ragen bei einer alpha-Helix immer nach außen, daher können auch Aminosäuren mit etwas größeren Seitenketten an einer alpha-Helix beteiligt sein. Ein großes Problem ist dabei die Aminosäure Prolin, die ja eine zyklische Struktur besitzt und keine "echte" Aminogruppe hat, und somit auch kein Amino-H-Atom, das eine H-Brücke bilden könnte.
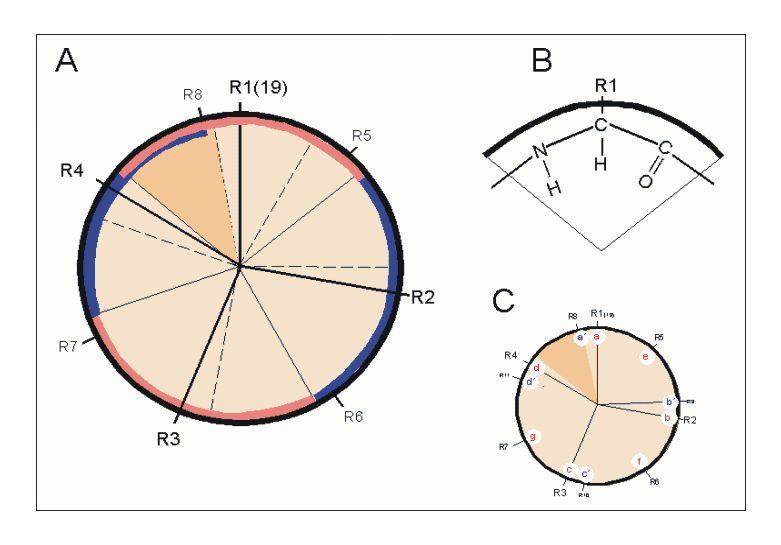
Eine alpha-Helix von oben betrachtet
Autor: Juergen Bode 2004, Lizenz: Creative Commons Attribution-Share Alike 3.0 Unported license.
Auf dieser Zeichnung aus der Wikipedia sieht man eine alpha-Helix von oben, die Aminosäuren wurden mit R1 bis R8 durchnummeriert. Man sieht sehr schön, dass Aminosäure R1 fast genau über R8 liegt. Die Aminosäure R19 ist hier nicht eingezeichnet, aber diese Aminosäure liegt exakt unter R1.
Die H-Brücke wird von R1 zu R4 ausgebildet, aber R5 bildet einen ähnlichen Winkel mit R1 wie R4. Dennoch sind Helix-Strukturen mit R1-R5-H-Brücken instabiler als Strukturen mit R1-R4-H-Brücken.
Aminosäuren in der alpha-Helix
Wie man sich vielleicht denken kann, sind nicht alle Aminosäuren für die Bildung einer solchen Helix geeignet. Die Eignung einer Aminosäure, eine alpha-Helix zu bilden, wird als Helixpotenzial Pα bezeichnet. Den Wert für dieses Helixpotenzial errechnet man aus der Häufigkeit, mit der eine Aminosäure in verschiedenen alpha-Helices auftritt.
Laut Stryer [3] können Verzweigungen in der Seitenkette (Valin, Threonin, Isoleucin) eine alpha-Helix sterisch destabilisieren, das Helixpotenzial dieser Aminosäuren sollte also verhältnismäßig klein sein. Eine Liste mit den Pα-Werten finden wir in dem Wikipedia-Artikel "Alpha-Helix" [1]. Danach hat die Glutaminsäure mit einem Pα von 1,59 das größte Helixpotenzial. Die zweit- und dritthäufigsten Aminosäuren sind Alanin (1,41) und Leucin (1,34) , gefolgt von Methionin (1,30), Glutamin (1,27), Lysin (1,23) und Arginin (1,21).
Schauen wir uns nun die Werte für die drei im Stryer genannten Aminosäuren an: Valin = 0,9, Threonin = 0,76 und Isoleucin = 1,09.
Diese Werte sind zwar deutlich kleiner als der von Glutaminsäure, Alanin oder Leucin, aber es gibt eine ganze Reihe von Aminosäuren, die noch seltener in alpha-Helices anzutreffen sind. Die geringste Häufigkeit hat die Aminosäure Prolin mit einem Pα-Wert von 0,34. Das liegt natürlich daran, dass Prolin keine freie Aminogruppe hat, sondern das N-Atom ist in einen Ring eingebaut. Befindet sich Prolin in einem Peptid, hat das N-Atom kein freies H-Atom mehr, so dass auch keine H-Brücke zum Carbonyl-O-Atom der viert nächsten Aminosäure gebildet werden kann. Prolin wird daher auch als "Helixbrecher" bezeichnet.
Aber auch Glycin (0,43) und Serin (0,57) neigen nicht gerade zur Bildung von alpha-Helices. Beim Glycin ist das etwas merkwürdig, weil es sich doch um die kleinste und einfachste Aminosäure handelt. Sterische Gründe für das seltene Vorkommen in der alpha-Helix sollten hier also keine Rolle spielen. Vielmehr ist es so, dass - gerade weil Glycin so klein ist - diese Aminosäuren dazu neigt, andere, noch stabilere Strukturen zu bilden als eine alpha-Helix [2].
alpha-Helices in Proteinen
Nicht jedes Peptid besitzt eine alpha-Helix, die meisten größeren Proteine enthalten aber zwei, drei oder noch mehr diese Helices. Allgemein kann man sagen, dass ungefähr jede vierte Aminosäure in einem Protein Bestandteil einer alpha-Helix ist [2]. Natürlich variiert dieser Anteil von Protein zu Protein. Ein Extrembeispiel ist das Ferritin, ein Eisenspeicherprotein. Hier liegt der Helixanteil bei sage und schreibe 75% [3, 4].

Oberflächenmodell des Maus-Ferritins
Autor: Vossmann, Lizenz: This work is free software; you can redistribute it and/or modify it under the terms of the GNU General Public License as published by the Free Software Foundation; either version 2 of the License, or any later version.
Auf diesem Bild sehen wir ein Modell des Maus-Ferritin-Komplexes. Dieser Komplex ist aus vielen Untereinheiten aufgebaut. Eine dieser Untereinheiten ist auf dem Bild violett hervorgehoben.
Faktoren, die eine alpha-Helix stabilisieren
Wie eben bereits gesagt, gibt es Aminosäuren, die recht häufig in einer alpha-Helix anzutreffen sind, während andere Aminosäuren recht selten oder im Fall von Prolin so gut wie gar nicht in einer solchen Helix vorkommen. Aber es gibt noch weitere Faktoren, die man berücksichtigen muss, wenn es um die Stabilität einer alpha-Helix geht.
Benachbarte Aminosäuren
Wechselwirkungen zwischen den Seitenketten von zwei benachbarten Aminosäuren können die Stabilität einer Helix spürbar beeinflussen. Als "Nachbar" der Aminosäure n gilt dabei nicht nur die Aminosäure n+1, die in der Primärstruktur auf Aminosäure n folgt, sondern auch die Aminosäure n+4, mit der die Aminosäure n eine H-Brücke bildet.
Saure und basische Aminosäuren
Handelt es sich bei den Aminosäuren n und n+4 beispielsweise um Glutaminsäure, dann sind bei einem pH-Wert von 7 die beiden COOH-Gruppen der Seitenketten negativ geladen und stoßen sich ab. Das destabilisiert dann die Helix. Das Gleiche passiert, wenn es sich bei diesen beiden Aminosäuren um basische Aminosäuren wie Lysin oder Arginin handelt. Bei einem pH-Wert von 7 sind die Seitengruppen positiv geladen und stoßen sich ab.
Umgekehrt ist es, wenn sich an Position n eine saure, an Position n+4 aber eine basische Aminosäure befindet. Die negativ bzw. positiv geladenen Seitengruppen ziehen sich an und stabilisieren die Helix.
Befindet sich an Position n eine Aminosäuren mit negativ geladener Seitengruppe und an Position n+2 oder n+3 eine Aminosäure mit negativer Seitengruppe, dann ziehen sich diese beiden Aminosäuren an und stören bzw. verbiegen die Helixstruktur.
Sterische Faktoren
Befinden sich an den Positionen n und n+4 zwei Aminosäuren mit sehr großen Seitengruppen, dann kommen sich die Atome dieser Seitengruppen "in die Quere", es liegt also eine sterische Behinderung vor, durch die die alpha-Helix destabilisiert wird.
Superhelices
Die Aminosäuren einer alpha-Helix können so verteilt sein, dass sich auf der einen Längsseite hauptsächlich Aminosäuren mit hydrophilen Resten befinden, auf der anderen Seite vorwiegend Aminosäuren mit hydrophoben Resten. Solche Helices spielen eine wichtige Rolle in bestimmten Proteinen der Zelle, vor allem bei Membranproteinen.
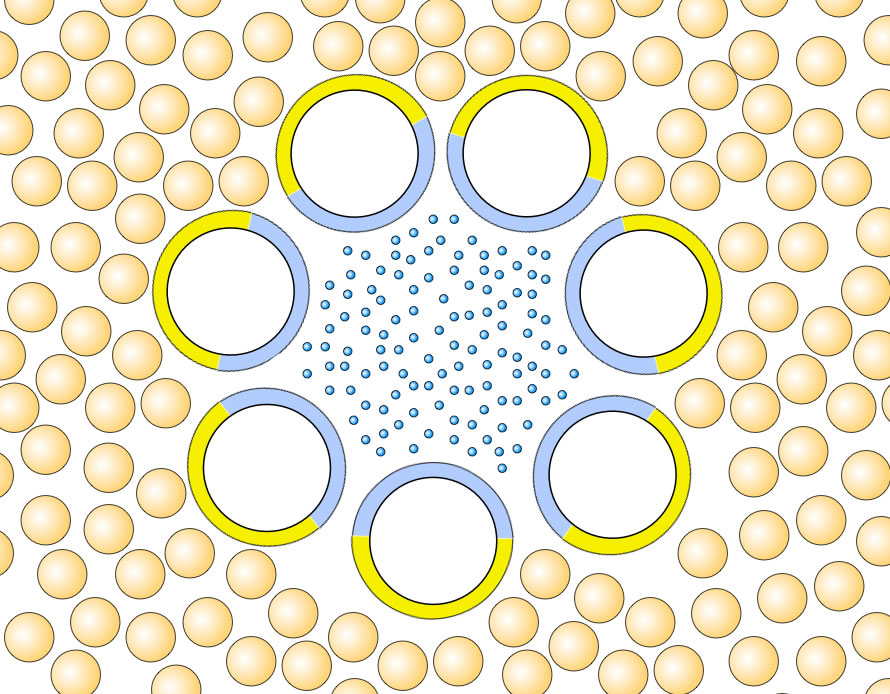
Sieben alpha-Helices bilden einen hydrophilen Kanal in der Zellmembran
Autor: Ulrich Helmich, Lizenz: Public domain
Jede der hier gezeigten alpha-Helices besitzt eine hydrophobe (gelb) und eine hydrophile (blau) Seite. Innerhalb des Membranproteins ordnen sich diese sieben Helices nun so an, dass die hydrophilen Seiten einander zugewandt sind und einen hydrophilen Kanal quer durch die Membran bilden, durch den Wasser-Moleküle und Ionen (blaue Kugeln) passieren können.
Oft kommen aber auch Superhelices aus drei, vier, fünf oder sechs solcher alpha-Helices vor, und nicht immer bilden solche Superhelices Kanäle in der Membran, sondern haben andere Aufgaben in der Zelle wie zum Beispiel das Rhodopsin, eines der Sehpigmente in der Netzhaut der Wirbeltiere.
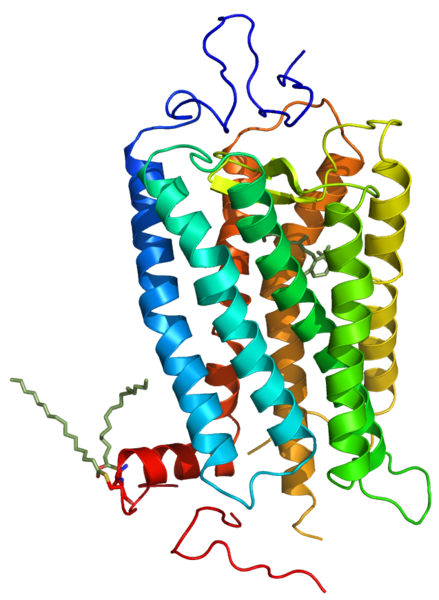
Das Membranprotein Rhodopsin besteht aus sieben alpha-Helices
S. Jähnichen, Public domain, via Wikimedia Commons
Auf dieser Seite der Zellbiologie-Abteilung erfahren Sie mehr über Membranproteine, deren Aufgaben und deren Struktur.
Die zweite wichtige Sekundärstruktur, die man in Peptiden und Proteinen findet, ist das beta-Faltblatt. Diese Struktur wurde kurz nach der alpha-Helix von Pauling und Corey entdeckt.
Quellen:
- Nelson, Cox. LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Wikipedia, Artikel "alpha-Helix"
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.
- Wikipedia, Artikel "Ferritin"