Schritt 1 der DNA-Kondensation
Die DNA-Doppelhelix in den Zellkernen eukaryotischer Lebewesen ist mit Hilfe von bestimmten Proteinen, den Histonen, stark kondensiert (siehe "Die Organisation der DNA"). Diese Kondensation wird in mehreren Schritten erreicht. Diese Webseite behandelt den ersten Schritt dieser DNA-Kondensation.
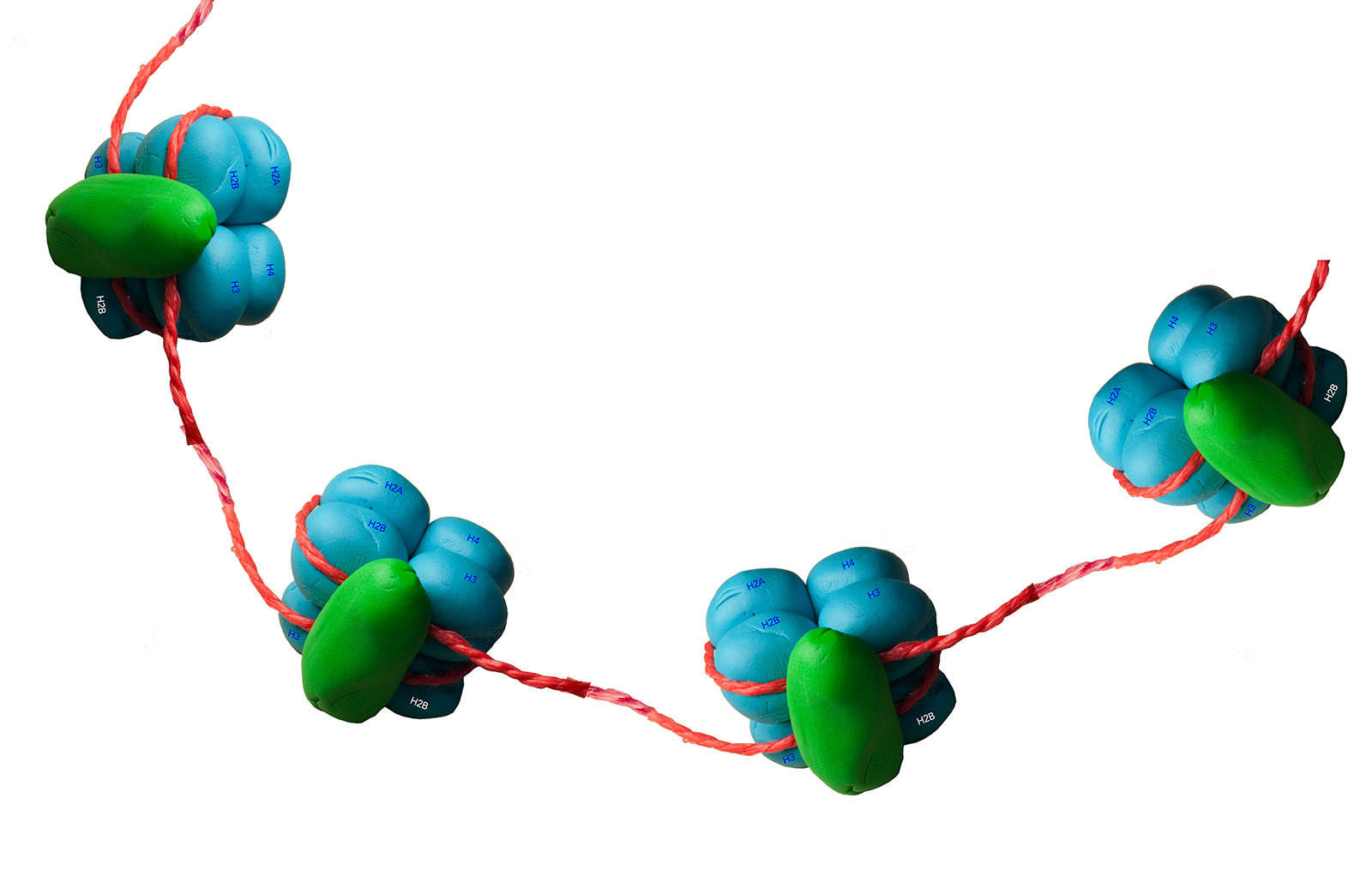
Die DNA windet sich um mehrere Nucleosomen
Autor: Ulrich Helmich 2020. Dieses Bild baut auf dem Bild von Darekk2 aus der Wikipedia auf, daher unterliegt es ebenfalls der "Creative Commons Attribution-Share Alike 3.0 Unported" Lizenz.
Auf diesem Bild, das auf den ersten Blick aussieht wie eine Perlenkette, sieht man einen Ausschnitt aus der DNA (roter Faden), die sich um mehrere Komplexe aus Histonen windet.
Histone sind Arginin und Lysin reiche basische Proteine. Die positiv geladenen NH3+-Seitenketten dieser Histone ziehen die negativ geladenen Phosphatgruppen der DNA-Doppelhelix elektrisch an.
Diese Komplexe aus 8 Histonen werden als Nucleosomen bezeichnet. Im folgenden Abschnitt wollen wir uns den Bau eines solchen Nucleosoms einmal näher anschauen.
Bau eines Nucleosoms

Organisation eines Nucleosoms (Fimo-Modell)
Autor: Ulrich Helmich 2021, Lizenz: Public domain.
Der Core-Komplex
Der sogenannte Core-Komplex eines Nucleosoms besteht aus acht Untereinheiten, die als H2A, H2B, H3 und H4 bezeichnet werden. Jedes Histon kommt zweimal in einem Nucleosom vor.
Die Histone H2A und H2B vereinigen sich bei der Bildung eines Nucleosoms zunächst zu H2A/B-Dimeren. Entsprechend bilden die Histone H3 und H4 die H3/H4-Dimere. Zwei H3/H4-Dimere bilden dann einen H3/H4 Tetramer (auf der Abbildung quasi die rechte Seite des Fimo-Modells). Jeder H3/H4-Tetramer vereinigt sich dann mit zwei H2A/H2B-Dimeren zu einem Octamer.
Der gesamte Core-Komplex hat einen Durchmesser von 10 bis 11 nm.
Die DNA
Die DNA wickelt sich ca. mit 145 bis 147 Basenpaaren [1,2,5] mit 14 internen Helixwindungen [5] ca. 1,6 bis 1,7 mal um den Komplex aus acht Histon-Molekülen.
Von den 14 Windungen der Doppelhelix haben mindestens 12 direkten Kontakt mit den Histonen des Nucleosoms [5].

Die DNA windet sich um den Core-Komplex aus acht Histonen
Zephyris at the English-language Wikipedia, CC BY-SA 3.0, via Wikimedia Commons
Auf diesem Bild aus der engl. Wikipedia sieht man sehr schön, wie sich die DNA um den aus acht Histonen bestehenden Core-Komplex windet. Interessant ist auch, dass die "Arme" der Histon-Moleküle aus dem Nucleosom herausragen. Diese langen Aminosäure-Ketten am NH2-Ende der Histone können leicht modifiziert werden, sie können Kontakt mit anderen Proteinen aufnehmen, und sie können auch Kontakt mit den Histonketten benachbarter Nucleosomen aufnehmen.
Histon H1
Das Histon H1 (in dem Fimo-Modell grün dargestellt) "verschließt" den Core-Komplex mit der DNA.
Anschaulich könnte man sich hier vorstellen, dass H1 das "Abrutschen" der DNA vom Core-Komplex verhindert.
Histon H4
Das Histon H4 spielt eine Rolle bei der Bildung der nächst übergeordneten Struktur. H4 hat sowohl am Carboxy- wie auch am Aminoende "Arme", die aus dem Komplex herausragen. Mit diesen Armen kann sich das Nucleosom mit den Nachbar-Nucleosomen verbinden, dazu nehmen die Arme Kontakt mit dem H2A/H2B-Dimer im Nachbar-Nucleosom auf.
Die Nucleosomen der "Perlenkette" werden also nicht nur durch den DNA-Faden zusammengehalten, sondern nehmen auch direkt untereinander Kontakt auf.
Acetylierung von H4
Das Histon H4 spielt noch eine weitere wichtige Rolle in den Nucleosomen. Vier Lysin-Reste im H4-Arm können von der Histon-Acetyl-Transferase acetylisiert werden. Das Enzym überträgt einen Acetylrest von Acetyl-Coenzym A auf die Aminogruppe von Lysin-Seitenketten. Durch die Acetylisierung werden positive Ladungen des Histons neutralisiert, und die DNA wird weniger stark an das Nucleosom gebunden, was wiederum die Transkription der DNA erleichtert [4].
Nucleosomen-Dichte der DNA
Wenn man sich in Biologie-Büchern die verschiedenen Abbildungen der Nucleosomen-Perlenkette anschaut, könnte man denken, dass die Nucleosomen gleichmäßig verteilt sind und dass die Abstände zwischen den Nucleosomen immer die gleiche Länge haben. Meistens werden Angaben wie 20 bis 60 Basenpaare für die Linker-DNA zwischen den Nucleosomen gemacht.
Dieses Bild täuscht aber. Der Fachbegriff der Nucleosomenpositionierung[5] beschreibt die Tatsache, dass sich die Nucleosomen an bestimmte DNA-Bereiche lieber bzw. häufiger binden als an andere DNA-Bereiche.
DNA-Abschnitte, bei denen auf dem einen Strang nur Purinbasen Adenin und Guanin vorkommen (logischerweise dann auf dem anderen Strang die komplementären Pyrimidinbasen Thymin und Cytosin) können Nucleosomen nicht so gut binden wie "normale" DNA-Abschitte, in denen die Basen gleichmäßiger verteilt vorkommen. Solche DNA-Sequenzen finden sich im Bereich des Centromers, aber auch im Promotorbereich einiger Gene. Durch die geringe Nucleosomen-Dichte in diesen Bereichen können Proteine wie zum Beispiel die RNA-Polymerase leichter an die DNA andocken, um ihre Aufgaben zu erfüllen [5] .
Quellen:
- Alberts, Bruce et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.
- Knippers, Molekulare Genetik, Stuttgart 2006.
- Spektrum-Lexikon der Biologie, Artikel "Histon-Acetylierung".
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.