Vorbemerkungen
Der Mensch, die Säugetiere und viele andere Tiere auch haben bekanntlich ein Immunsystem, das eingedrungene Krankheitserreger unschädlich macht. Wenn eine bakterielle oder virale Infektion dann überstanden ist, werde Spuren der Fremd-DNA in unserem Immunsystem in speziellen Zellen archiviert. Damit ist des dem Immunsystem möglich, bei einer erneuten Infektion mit dem gleichen Erreger wesentlich schneller zu reagieren als bei der Erstinfektion (Immungedächtnis).

Berge auf den Färöer-Inseln
Autor: Martin Helmich 2022, Lizenz: siehe Seitenende
Ein spektakuläres Beispiel für das Immungedächtnis ist eine Masern-Epidemie auf den Färöer-Inseln (siehe Abb. 1). 1781 wurde die abgelegene Inselgruppe von den Masern heimgesucht, viele Einwohner, vor allem viele Kinder erkrankten daran. 1846 kam es zu einem erneuten Masern-Ausbruch. Von den älteren Einwohnern, die bereits 1781 erkrankt waren, erkrankte keiner zum zweiten Mal, aber viele von den jüngeren Einwohnern erwischte die Krankheit [1].
Das Immungedächtnis der Prokaryoten
Wer hätte es gedacht - auch Bakterien und Archaeen haben ein Immungedächtnis. DNA-Sequenzen eingedrungener Viren werden in bestimmten Abschnitten der prokaryotischen DNA archiviert. Bei Bedarf werden diese archivierten DNA-Abschnitt in kurze mRNAs transkribiert, und diese mRNAs leiten dann Endonucleasen zu der eingedrungenen Viren-DNA. Die Archiv-mRNA paart sich dann mit dem komplementären Abschnitt der Viren-DNA, und die mitgebrachte Endonuclease zerschneidet die Viren-DNA (oder Viren-RNA) und macht diese so unschädlich.
Schauen wir uns jetzt einmal an, wie dieses Abwehrsystem genau funktioniert.
Das Archiv der Prokaryoten
Autor: Ulrich Helmich 2022, Lizenz: siehe Seitenende
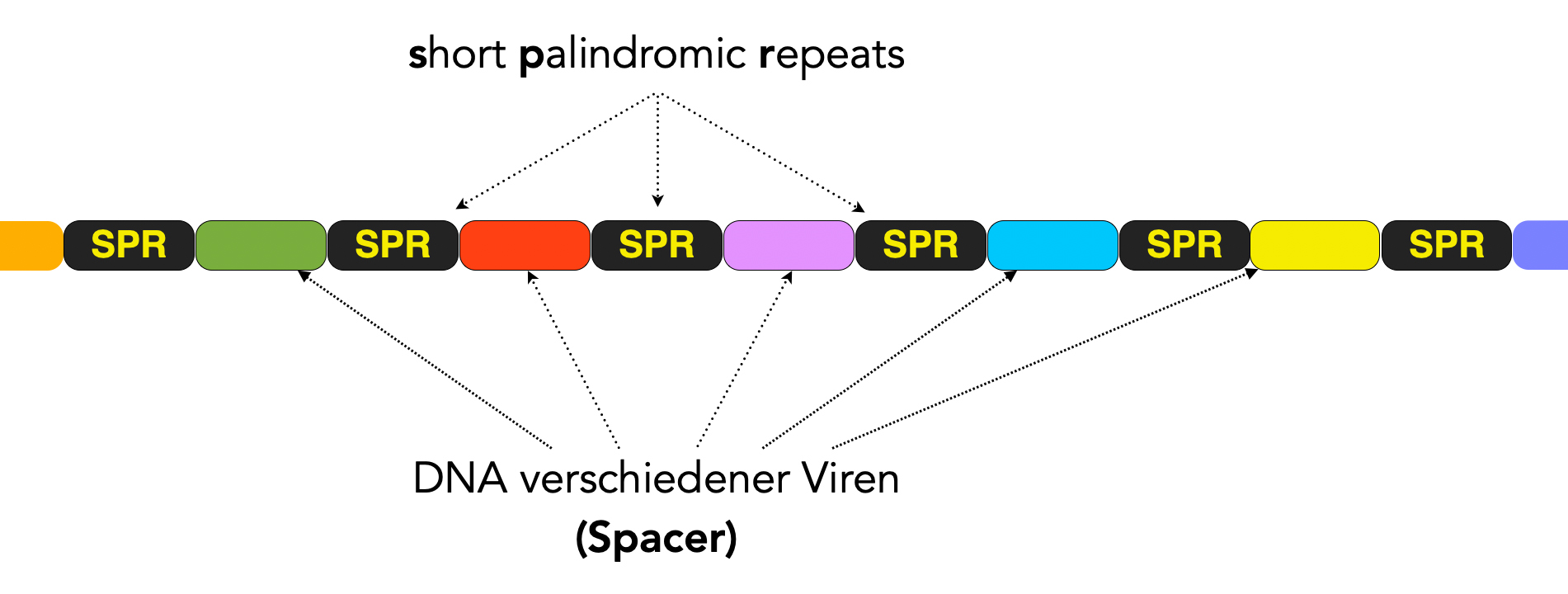
Das "Bücherregal" des prokaryotischen Viren-Archivs besteht aus ca. 23-55, im Schnitt 32 Basenpaaren langen palindromischen Wiederholungs-Einheiten (engl.: short palindromic repeats, kurz SPR) [2, 3].
Eine palindromische DNA-Sequenz liest sich von hinten genau so wie von vorne. Hier ein Beispiel: ACCTGCTTCGTCCA. In der deutschen Sprache wäre beispielsweise "Anna" ein solches Palindrom, oder der Satz "Eine Horde bedrohe nie".
Man kennt heute über 40 verschiedene Sequenzmotive für diese Repeats, über 30 dieser palindromischen Sequenzen weisen Haarnadel-Sekundärstrukturen auf [3].
Die "Bücher" in diesem Regal bestehen dann aus kurzen Abschnitten (21-72 Basenpaare) der "eingefangenen" Viren-DNA. In der Fachsprache bezeichnet man diese Viren-Abschnitte als Spacer.
Diese SPRs mit den eingeschobenen Spacern treten bei 90% der Archaeen und 40% der Bakterien gehäuft auf und werden daher als "gehäuft auftretende, regelmäßig unterbrochene, kurze Palindrom-Wiederholungen" bezeichnet. Dies ist die Übersetzung der englischen Formulierung "Clustered Regularly Interspaced Short Palindromic Repeats". Diesen kaum aussprechbaren Satz kürzt man üblicherweise mit CRISPR ab. Die Anzahl der Wiederholungseinheiten "liegt zwischen einigen wenigen zum zu mehreren hundert pro CRISPR-Locus" [3].
Der CRISPR-Abschnitt in der DNA der Prokaryoten
Autor: Ulrich Helmich 2022, Lizenz: siehe Seitenende
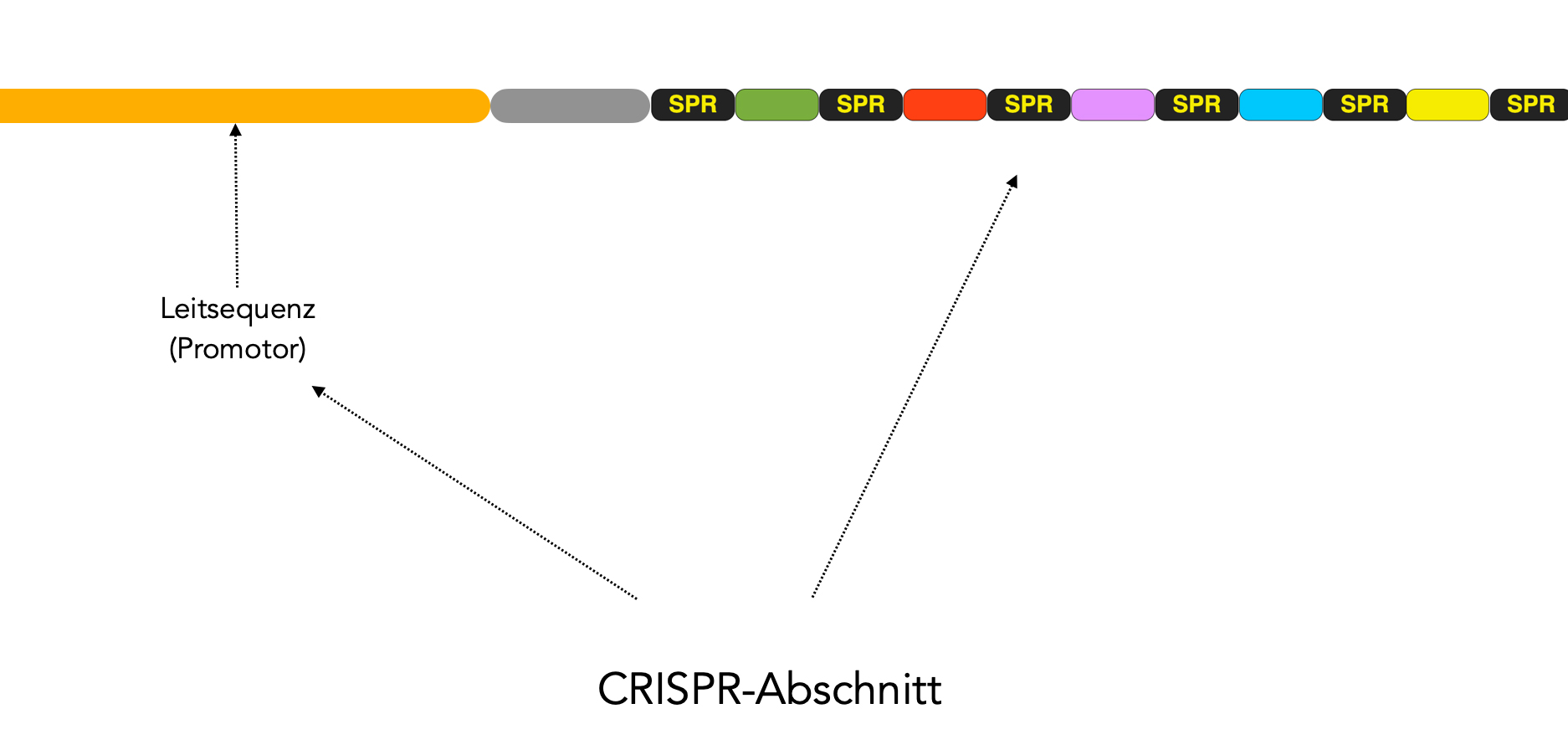
Vor diese eigentliche CRISPR-Sequenz ist noch eine 200 bis 250 Basenpaare lange Leitsequenz geschaltet, die als eine Art Promotor/Operator dient. Verschiedene regulatorische Enzyme können an diese Leitsequenz andocken und die Transkription der stromabwärts gelegenen SPR/Spacer-Einheiten regulieren.
Der gesamte Bereich aus Leitsequenz und den short palindromic repeats mit zwischengeschalteten Spacern wird als CRISPR-Abschnitt bezeichnet [2].
Entdeckt wurde das CRISPR-System bereits 1987 in Escherichia coli-Bakterien von Yoshizumi Ishino et al. [4] an der Osaka Universität in Japan. Allerdings hatten die Forscher damals noch keine Ahnung, was sie da tatsächlich entdeckt hatten.
Damit hätten wir das Immungedächtnis der Prokaryoten kurz beschrieben. Zwei Fragen sind allerdings noch nicht geklärt.
- Wie wird die fremde Viren-DNA erkannt, ausgeschnitten und in den CRISPR-Abschnitt hinein gepackt?
- Wie erfolgt die "Auswertung" der Viren-Bibliothek, um neu eingedrungene Viren unschädlich zu machen?
Diese beiden Fragen wollen wir in den nächsten Abschnitten beantworten.
1. Einbau neuer Viren-DNA
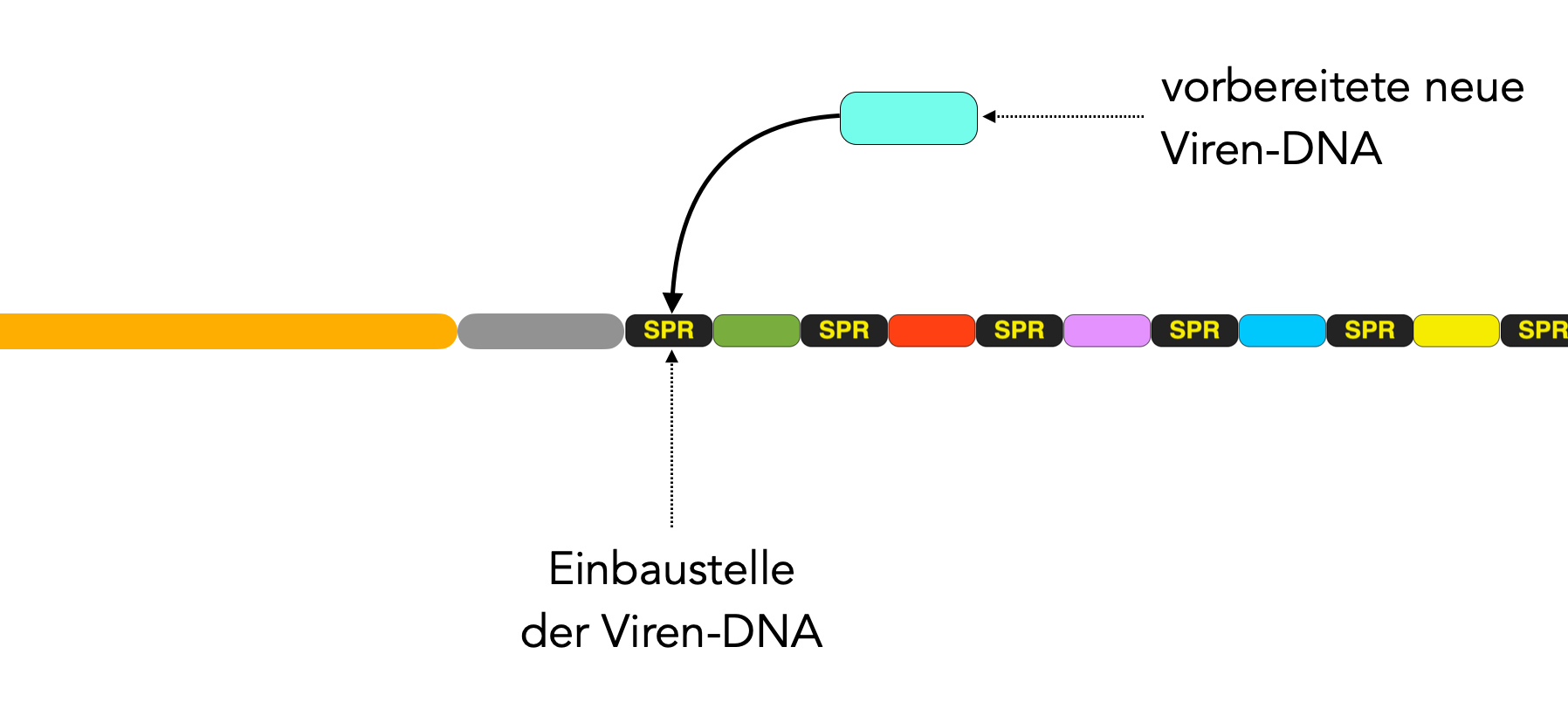
Der CRISPR-Cas-Mechanismus der Prokaryoten ist in drei Schritte unterteilt. Der erste Schritt, die Adaptation, besteht in dem Erkennen fremder Viren-DNA und dem Einbau derselben in die CRISPR-Region des bakteriellen Genoms.
Die eingedrungene Viren-DNA wird durch Endonucleasen gespalten und durch verschiedene Cas-Enzyme (Cas1 und Cas2 [5] bzw. Cas1 und Cas7 [2]) weiter verarbeitet. So vorbereitet, wird sie dann direkt in die erste Repeatsequenz hinter die Leitsequenz eingebaut:
Für Experten:
Einbau der Fremd-DNA
Den Ablauf dieses Einbaus stellt man sich ungefähr folgendermaßen vor [3]:
- Das erste SPR-Element hinter der Leitsequenz wird in zwei Einzelstränge zerlegt (vereinfachte Darstellung; ganz so einfach ist es nicht...).
- Die neue Viren-DNA wird nun zwischen die 5'-Enden der beiden Einzelstränge "eingehängt".
- Durch DNA-Replikation werden die beiden Einzelstränge zu zwei neuen SPR-Elementen vervollständigt.
Natürlich sind einige Enzyme an diesen Vorgängen beteiligt, die hier nicht berücksichtigt worden sind.
2. Vernichtung der Viren-DNA/RNA
Wir werden nun die in der CRISPR-Bibliothek gespeicherten Informationen ausgewertet, um eingedrungene Viren zu bekämpfen?
Wir müssen uns den Aufbau des Prokaryoten-Genoms noch genauer anschauen:
Aufbau der CRISPR-Cas-Region
Autor: Ulrich Helmich 2022, Lizenz: siehe Seitenende
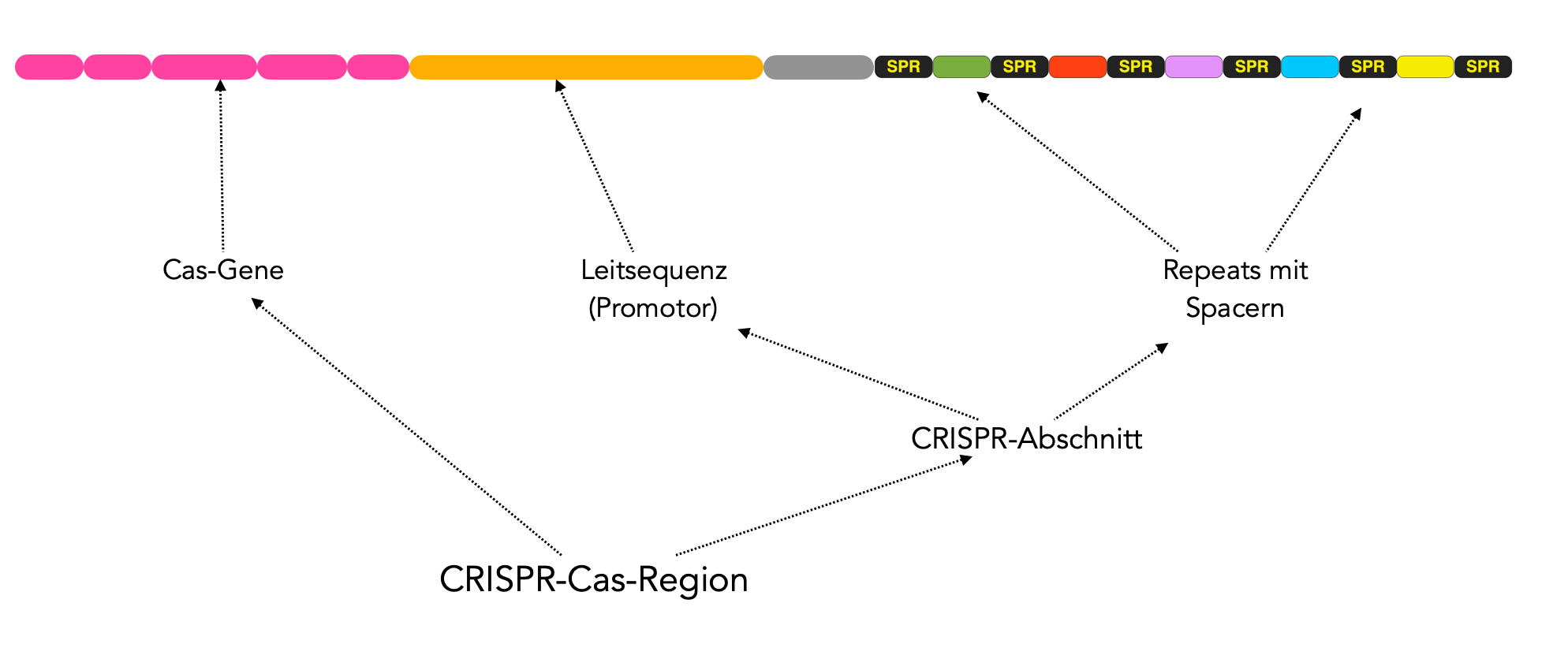
Stromaufwärts, also vor der CRISPR-Leitsequenz, befindet sich eine Reihe von Genen für Enzyme, mit denen eingedrungene Viren bekämpft werden. Diese Enzyme befinden sich immer vor dem CRISPR-Abschnitt, sind sozusagen mit ihm assoziiert. Im Englischen bezeichnet man diese Gene als "CRISPR-associated genes", was dann mit "Cas" abgekürzt wird. Daher erklärt sich auch die Bezeichnung "CRISPR-Cas".
Kommen wir nun zur Frage, wie dieses CRISPR-Cas-System eingedrungene Viren bekämpft.
Die RNA-Polymerase setzt sich an die Leitsequenz des CRISPR-Bereichs und synthetisiert eine lange mRNA, die eine Kopie des gesamten CRISPR-Bereichs ist, also alle SPR- und dazwischenliegende Spacer-Elemente besitzt. Diese mRNA wird als pre-crRNA bezeichnet.
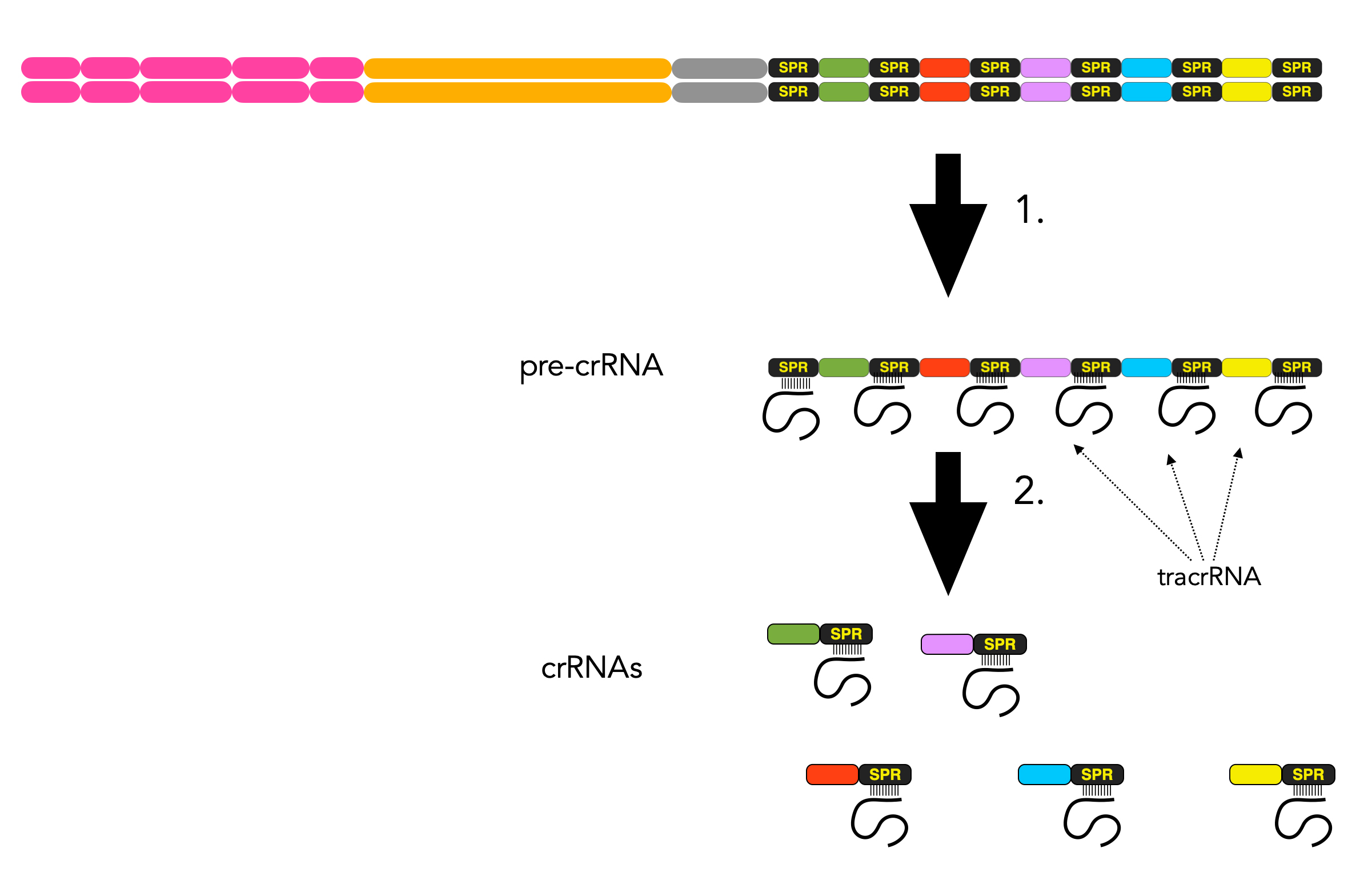
Transkription der CRISPR-Region (1) und Zerlegung der pre-crRNA in crRNAs (2)
Autor: Ulrich Helmich 2022, Lizenz: siehe Seitenende
An die Repeats der pre-crRNA lagern sich nun kurze komplementäre RNA-Stücke an, die sogenannten Trans-activating crRNA (kurz tracrRNA). Dann wird diese pre-crRNA durch das Enzym RNAase III in einzelne crRNAs zerlegt. Jede crRNA enthält einen Spacer mit einer Viren-Sequenz und einen Teil der Wiederholungs-Einheiten mit der daran gebundenen tracrRNA [6].
Nun kommt ein besonderes Cas-Enzym ins Spiel, das Enzym Cas9. Dieses Enzym ist eine Endonuclease, kann also DNA in der Mitte durchtrennen (während Exonucleasen die Enden der DNA auflösen). Allerdings darf das Enzym nicht einfach so (unkontrolliert) freigesetzt werden, dann würde es ja sämtliche DNA der Prokaryotenzelle zerschneiden. Nein, es muss spezifisch zu der Stelle geleitet werden, die es zerschneiden soll.
Diese Lotsen-Aufgabe übernehmen nun die crRNAs, die wir gerade betrachtet haben. Eine crRNA setzt sich dabei zusammen mit ihrer tracrRNA in eine Tasche des Cas9-Enzyms.
Diese Komplexe (siehe Bild unten) patrouillieren dann durch die Prokaryotenzelle [8], bis sie zufällig mit einer DNA oder RNA in Kontakt kommen. Sollte die Basensequenz dieser Nucleinsäure zufällig komplementär zu der Tracer-Sequenz des Komplexes sein, bindet die Tracer-RNA komplementär an diese DNA bzw. RNA. Das Cas9-Enzym zerschneidet die Nucleinsäure dann wie eine Schere.
Da die Spacer nur DNA-Sequenzen von eingedrungenen Viren enthalten, kann auf diese Weise auch nur Viren-DNA oder -RNA unschädlich gemacht werden.
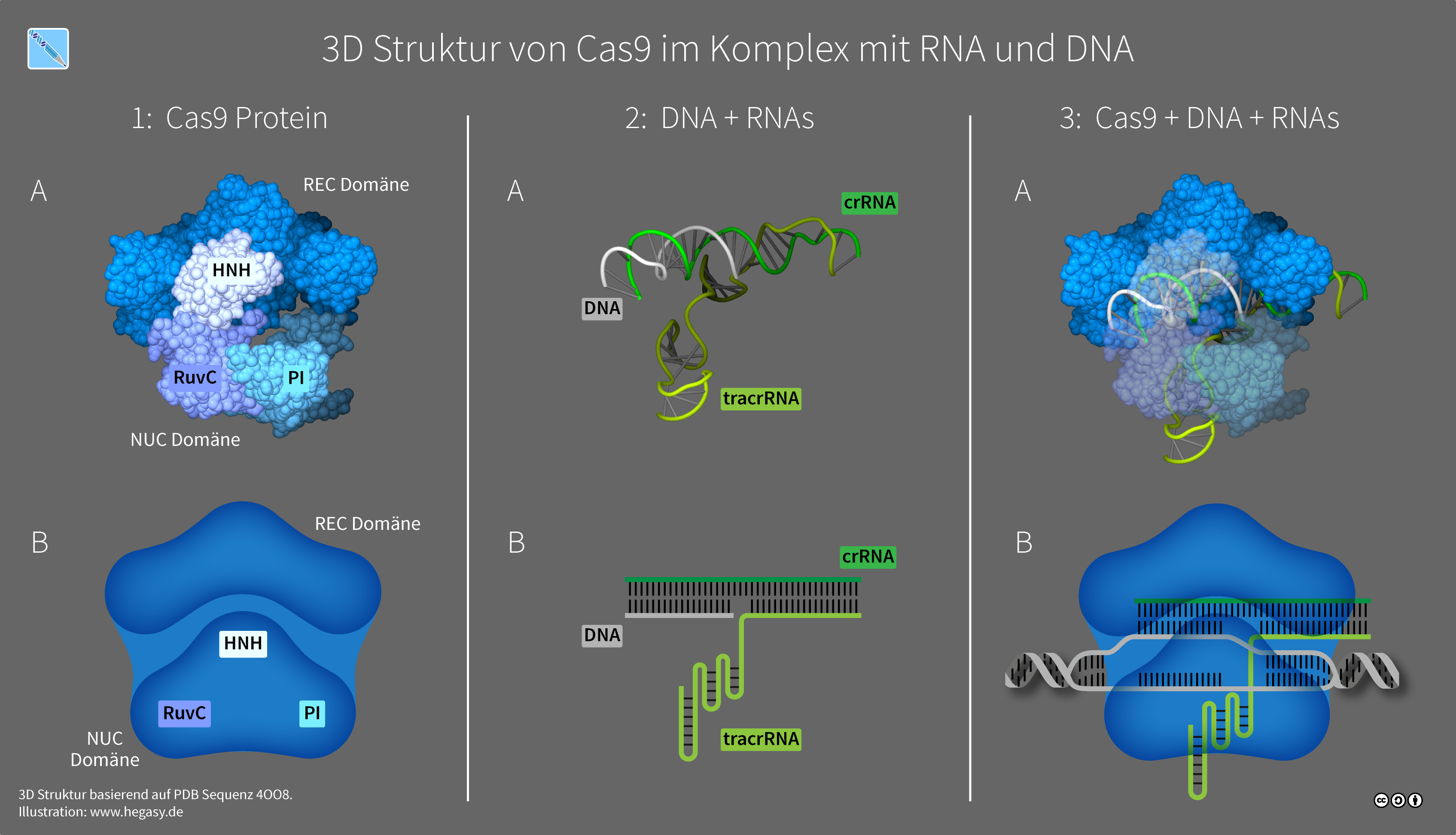
Der Cas9-DNA-RNA-Komplex
Guido4, CC BY-SA 4.0, via Wikimedia Commons
Dieses Bild aus der Wikipedia [7] zeigt sehr schön den Aufbau dieses Komplexes aus dem Enzym Cas9, der crRNA/tracrRNA und einem DNA-Stück, das durchtrennt werden soll.
Quellen:
- Schütt, Bröker, Grundwissen Immunologie, 2. Auflage, Springer-Verlag 2009.
- Nordheim, Knippers: Molekulare Genetik, 11. Auflage, Thieme-Verlag Stuttgart 2018.
- Maier: Das CRISPR-Cas-System von Haloferax volcanii, Dissertation, Universität Ulm 2014.
- Wikipedia, Artikel "CRISPR".
- Klein et al.: "CRISPR-Cas-Systeme der Klasse 1: Genome Engineering und Silencing", BIOspektrum 04.22
- Max-Planck-Gesellschaft, Artikel "CRISPR-Cas9 - eine Schere aus Enzym und RNA", kein Datum, kein Autor angegeben.
- Wikipedia, Artikel "CRISPR/Cas9-Methode".
- Spektrum direkt, Artikel "Bakterien-Immunabwehr mit Gedächtnis" vom 5. November 2010.